細菌プラスミドは宿主染色体とは独立に自律複製を行なう寄生性環状DNAであり、DNA複製の最も簡単なモデルシステムとして活発な研究がなされてきた。大腸菌プラスミドは多数知られており、それらは2種類のプラスミドが同一宿主内で安定に共存できない現象(不和合性能incompatibility:略してInc.)によって主として分類されている。また自律複製に必要な領域の塩基配列が明らかとなり、複製制御のメカニズムに関する知見も飛躍的に蓄積されてきた。これらの研究はたがいに異なった不和合性能グループに属するプラスミドが共通の祖先からどのように形成され多様化してきたかについての解析をも可能にしている。 ホモロジー検索の結果、IncFIグループに属するP307は、複製に必須なタンパク質をコードする複製構造領域がColIb-P9(IncI )と高い類似性を示すのに対し、複製タンパク質の発現を制御する領域はR100(IncFII)と高い類似性を示すことを見出した。一方、IncZに属するpMU2200の複製制御領域はColIb-P9に類似しているが、複製構造領域はR100と高い類似性を示すことが報告された。ColIb-P9とR100との間の塩基配列の類似性は40%程度で相同性は乏しい。これらのことは、ColIb-P9/R100あるいはP307/pMU2200のプラスミドが、P307/pMU2200あるいはColIb-P9/R100の先祖型の複製制御領域を互いに組換えることによって多様化してきたことを示している。しかし、今日にいたるまで組換えの性質については全く明らかにされていない。そこで私はまず、IncI )と高い類似性を示すのに対し、複製タンパク質の発現を制御する領域はR100(IncFII)と高い類似性を示すことを見出した。一方、IncZに属するpMU2200の複製制御領域はColIb-P9に類似しているが、複製構造領域はR100と高い類似性を示すことが報告された。ColIb-P9とR100との間の塩基配列の類似性は40%程度で相同性は乏しい。これらのことは、ColIb-P9/R100あるいはP307/pMU2200のプラスミドが、P307/pMU2200あるいはColIb-P9/R100の先祖型の複製制御領域を互いに組換えることによって多様化してきたことを示している。しかし、今日にいたるまで組換えの性質については全く明らかにされていない。そこで私はまず、IncI ファミリー、IncFIIファミリーの複製制御機構の解析をColIb-P9、R100を用いて行い、ついでColIb-P9、R100を出発材料にしてP307を模したキメラレプリコンを作製して、複製可能なキメラプラスミドが形成されるために必要な条件を決定することを試みた。 ファミリー、IncFIIファミリーの複製制御機構の解析をColIb-P9、R100を用いて行い、ついでColIb-P9、R100を出発材料にしてP307を模したキメラレプリコンを作製して、複製可能なキメラプラスミドが形成されるために必要な条件を決定することを試みた。 ColIb-P9はコピー数1-2の低コピープラスミドで、複製最小領域は約1.8kbである。この領域には複製起点(ori)と、複製の開始反応に必須なタンパク質をコードするrepZ遺伝子、repZ遺伝子の発現量を正、負にそれぞれ制御するrep Y、inc遺伝子が存在する(図1)。RepZはoriに作用して複製を開始すると考えられている。repYはrepZの5’端と10塩基重複しており、29アミノ酸からなるリーダーペプチドをコードしている。repZ、repYはオペロンを構成しており、これらの遺伝子に対応するmRNAはRepZ mRNAと名付けられている。inc遺伝子はrepY遺伝子の上流に位置し、RepZ mRNAとは逆向きに転写される72ヌクレオチドからなるアンチセンスIncRNAをコードする。ColIb-P9のコピー数を反映している複製開始頻度はRepZの合成量によって決定されており、それ故、repY、inc遺伝子作用によるrepZ遺伝子の発現調節が複製を制御している。 repZ発現の調節にはRepZ mRNAのとる二次構造の変化が重要であることが明らかとなっている。RepZ mRNAはinc、repYがコードされている領域において、すくなくとも3ヶ所で、それぞれ構造I、II、IIIと名付けられたステム・ループ構造を形成し(図1)、このうちrepZ発現の調節に関わるものは構造Iと構造IIIである。構造IはIncRNAのコードされる領域内に存在しており、IncRNAの標的である。構造IIIはrepZの翻訳開始シグナル(リボゾーム結合部位;S.D.配列と開始コドン)を被っており、repZの翻訳開始を抑えている。repZの翻訳には構造IIIがrepYの翻訳に共役して破壊されるとともに、構造IIIステムに存在する5’CGCC3’配列が、107塩基上流の構造Iルーブに存在する相補的な5’GGCG3’配列とまず対合し、つぎに両配列の近傍6塩基ずつがさらに対合して計10塩基対からなるシュードノットと呼ばれる新たな二次構造を一過的に形成することが必要である。また、シュードノットの形成にはrepYの翻訳停止位置が極めて重要であり、その終止コドンの位置をRepZのアミノ酸配列を変化させることなしに30塩基下流に移すとrepZ発現は大きく低下することも明らかにされている。  図1 ColIb-P9プラスミドの複製領域の遺伝子構成と制御領域に予想されるRNA二次構造。a.ColIb-P9の複製領域の一次構造を模式的に示した。太い直線は最小複製領域を示す。長方形は複製に関与する遺伝子の位置を示している。oriはそれぞれの複製開始点を示す。横方向の直線は主な転写物を示している。+,-はrepZ、repA1発現がそれぞれ正負に制御されることを示す。塩基番号はBglII部位を1として表示した。b.RepZ mRNAの先導領域で明らかにされているRNA二次構造を示した。数字は塩基番号を示す。repY、repZの開始コドンと終止コドンは二重下線で示した。予想されるS.D.配列は長方形で囲んだ。incのプロモーター配列-10、-35領域は下線で示した。シュードノットに関与する相補性塩基配列は傍点で示した。矢印は塩基置換を示し、括弧内に変異名を記した。構造I、II、IIIは(I)、(II)、(III)で示した。 図1 ColIb-P9プラスミドの複製領域の遺伝子構成と制御領域に予想されるRNA二次構造。a.ColIb-P9の複製領域の一次構造を模式的に示した。太い直線は最小複製領域を示す。長方形は複製に関与する遺伝子の位置を示している。oriはそれぞれの複製開始点を示す。横方向の直線は主な転写物を示している。+,-はrepZ、repA1発現がそれぞれ正負に制御されることを示す。塩基番号はBglII部位を1として表示した。b.RepZ mRNAの先導領域で明らかにされているRNA二次構造を示した。数字は塩基番号を示す。repY、repZの開始コドンと終止コドンは二重下線で示した。予想されるS.D.配列は長方形で囲んだ。incのプロモーター配列-10、-35領域は下線で示した。シュードノットに関与する相補性塩基配列は傍点で示した。矢印は塩基置換を示し、括弧内に変異名を記した。構造I、II、IIIは(I)、(II)、(III)で示した。 一方、IncFIIファミリーに属するR100はコピー数1-2のプラスミドで、最小複製領域は1.86kbであり、この領域に複製起点ori、複製開始タンパク質遺伝子repA1、repA1の発現を転写レベル、翻訳レベルでそれぞれ負に制御するrepA2、inc遺伝子が存在することが明らかにされている。RepA1はrepA1の下流に存在するoriに結合することによって複製が開始されることが明らかにされている。 repA2はrepA1の上流約550塩基より翻訳開始される11kdからなるDNA結合タンパク質で、repA2のすぐ下流に存在するオペレーターに結合し、repA1をコードするmRNA(RNA-A)の転写を抑制する。incはrepA2とrepA1との間にコードされており、91塩基からなるRNA(RNA-E)を産生する。RNA-EはrepA1遺伝子のmRNAとは逆向きに転写され、mRNAと二重鎖を形成することによってrepA1の翻訳を阻害すると考えられている。 ColIb-P9とR100/R1の複製機構を比較すると、いずれのプラスミドも最小複製領域に複製タンパク質遺伝子をコードしており、その発現量が複製頻度を決定していること、oriが複製タンパク質遺伝子の下流に存在すること、いずれの複製タンパク質の発現も複製タンパク質遺伝子の上流にコードされるアンチセンスRNAによって抑制されていることの3点は共通である。一方、ColIb-P9において、R100/R1に同定されている転写レベルでのコピー数抑制因子が存在するかどうかは不明であり、またR100/R1においては、ColIb-P9のrepYに見られるリーダーペプチドが存在するかどうかは不明であった。 私はまずColIb-P9において、自律複製能を持ち、コピー数を野生型とほぼ同じに保つ領域を明らかにした。その結果、複製にはrepZ、repY、incを含む1758bpで十分であることを見出し、さらにRepZ mRNAの転写開始点よりincまでの全てのORFを欠失してもコピー数は野生型と同等に保たれることより、この領域にはrepA2に相当するコピー数抑制因子は存在しないと結論した。 次に、R100にもColIb-P9と類似した、repA1と5塩基オーバーラップする24アミノ酸からなるORF(ORF24)が存在していることに着目し、このORFに塩基置換を導入して複製能を検討した。ORF24の第4コドン目あるいは第12コドン目にアンバー変異を導入したプラスミド、開始コドンATGをACGに置換したもの、及び、終止コドンTGAをAGAに変換することにより、野生型と比べて60塩基下流で翻訳終結するものなどをそれぞれ作製してそれらの複製能とコピー数を測定した。いずれの変異に対しても複製能は失われたが、アンバー変異を導入したプラスミドをアンバーサプレッサ株(C600 supE)に導入したところ、複製能は回復した(表1)。また、アンバー変異、終止コドン変異体はいずれもrepA1の発現量が低下していた。以上のことから、ORF24は翻訳され、かつrepA1の開始コドン近傍で翻訳終結することがrepA1の発現、ひいては複製に必須であることが明らかになったので、このORFをrepA6と命名した。repA6がコードされる領域にはrepA1の開始コドン、S.D.配列を被うステム・ループ構造が存在し、この構造を破壊するような変異が導入されるとrepA1の発現がinc、repA6の制御を受けにくくなることが報告され、R100もColIb-P9と同様、アンチセンスRNAとリーダーペプチドからなる共通の制御モチーフを持ち、リーダーペプチドはrepA1の翻訳開始シグナルを被うRNA二次構造を破壊することが重要であることが示唆された(図2)。  表1 24アミノ酸からなるrepA6の複製に与える影響 表1 24アミノ酸からなるrepA6の複製に与える影響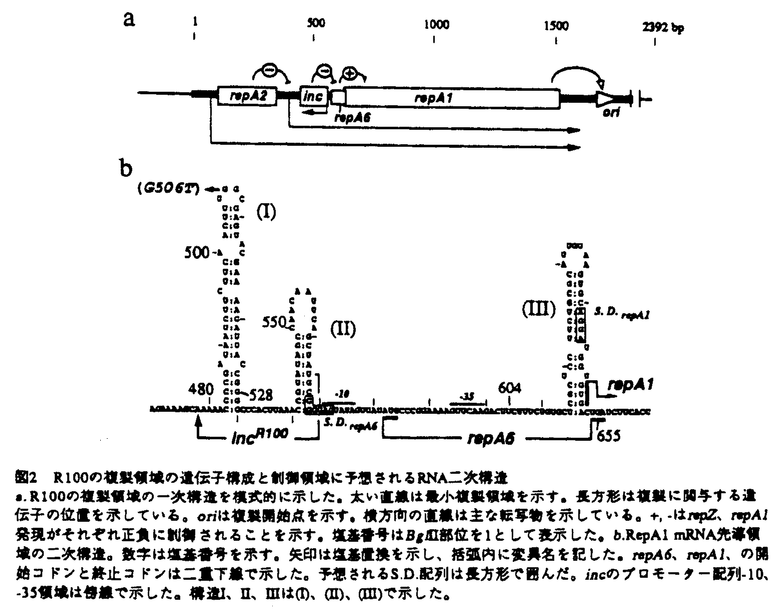 図2 R100の複製領域の遺伝子構成と制御領域に予想されるRNA二次構造a.R100の複製領域の一次構造を模式的に示した。太い直線は最小複製領域を示す。長方形は複製に関与する遺伝子の位置を示している。oriは複製開始点を示す。横方向の直線は主な転写物を示している。+,-はrepZ、repA1発現がそれぞれ正負に制御されることを示す。塩基番号はBglII部位を1として表示した。b.RepA1 mRNA先導領域の二次構造。数字は塩基番号を示す。矢印は塩基置換を示し、括弧内に変異名を記した。repA6、repA1、の開始コドンと終止コドンは二重下線で示した。予想されるS.D.配列は長方形で囲んだ。incのプロモーター配列-10、-35領域は傍線で示した。構造I、II、IIIは(I)、(II)、(III)で示した。 図2 R100の複製領域の遺伝子構成と制御領域に予想されるRNA二次構造a.R100の複製領域の一次構造を模式的に示した。太い直線は最小複製領域を示す。長方形は複製に関与する遺伝子の位置を示している。oriは複製開始点を示す。横方向の直線は主な転写物を示している。+,-はrepZ、repA1発現がそれぞれ正負に制御されることを示す。塩基番号はBglII部位を1として表示した。b.RepA1 mRNA先導領域の二次構造。数字は塩基番号を示す。矢印は塩基置換を示し、括弧内に変異名を記した。repA6、repA1、の開始コドンと終止コドンは二重下線で示した。予想されるS.D.配列は長方形で囲んだ。incのプロモーター配列-10、-35領域は傍線で示した。構造I、II、IIIは(I)、(II)、(III)で示した。 終止コドンは、導入した変異によってできたものの配列を示す。括弧内の数値はrepA1の開始コドンからrepA6の終止コドンまでの数を示す。Tcs形質転換体の割合は、検討したカナマイシン耐性(Kmr)の形質転換体のうちどれだけの割合がチトラサイクリン耐性(Tcr)になったかを示したものである。pACYC184(Tcr)のEcoRI部位に変異を導入したR100ミニレプリコン(Tcs)を挿入して得られたpWF1プラスミドをEcoRIで処理し、カナマイシン耐性遺伝子を含む1.3kbのEcoRI-EcoRI断片とライゲーションした。この反応液中には、R100断片にKmr断片が挿入された分子種とpACYC184にKmr断片が挿入された分子種が含まれる。この反応液をMC1061に直接導入して、カナマイシンブレート上で選択し、得られたカナマイシン耐性形質転換体を100コロニー前後とって、チトラサイクリン耐性の有無を調べた。もしR100断片が複製可能ならば、有意な数のTcs形質転換体が得られるはずである。調べたKmr形質転換体の総数のうち、Kmr、Tcsの形質転換体の割合を算出し、この値が0であれば複製不能、0よりも大きければ複製可能と判定した。 P307複製領域の塩基配列は、ori-repZ領域ではColIb-P9と90%、repZよりも上流部分で複製制御を担っている領域ではR100と88%の類似性を示している。また類似性がR100タイプからColIb-P9タイプに切り替わる境目は、repA6終止コドンの直後のrepZ遺伝子領域内であった。このことに着目して、上流にR100、下流にColIb-P9をもつキメラプラスミドを試験管内で作製し、複製能を検討した。R100のrepA6がColIb-P9のrepZと8bp重複するようにデザインされたキメラレプリコンpRC1(図3a)はR100と非常によく似たコピー数(R100が2.3コピーに対し、pRC1は2.5コピー)で安定に複製することを見いだした(表2)。このことは2つの異なったプラスミドの複製領域を組換えて機能的なレプリコンが新たに作製できることを示している。pRC1のrepA6にアンバー変異、開始コドン変異及び、終止コドン変異を導入したとき複製能は著しく阻害され、また不和合性能検定の結果、IncFIIグループに属することが明らかとなり、pRC1はR100と同じ複製制御を行うことが示された。 一方、repYとrepA6とのフレームを合わせて接続したプラスミドpRC3や、inc遺伝子内で組換えたプラスミドpRC2はいずれも複製しなかった(表2)。pRC2、pRC3の構造Iを構成する塩基をシュードノット形成が可能となるColIb-P9の配列に変えたpRCC2、pRCC3(図3b)は複製が可能となり、また、シュードノットができなくなる変異をpRCC2に導入した結果、複製は不能となった(表2)。これらの結果はColIb-P9の複製制御システムはシュードノットが維持されない限り複製不能となることを示している。  図3 a.in vitroで作製したキメラプラスミドpRC1、pRC2、pRC3の構造を模式的に示した。ColIb-P9の配列は白抜きの長方形で、R100の配列は影を付けた長方形で示した。縦の矢印はRNAの二次構造I、II、IIIの位置を示す。Pはプロモーターを示す。pRC1はR100のrepA1遺伝子の2番目のコドンをColIb-P9のrepZの5番目のコドンと接続して作製した。pRC2はR100のrepA6の7番目のコドンをColIb-P9のrepYの17番目のコドンと接続して作製した。pRC3はR100のG-528をColIb-P9のG-351に接続して作製した。b.ColIb-P9、R100、pRCC2、pRCC3の構造I上端部分の二次構造。ColIb-P9に由来する塩基は大文字で、R100に由来する塩基は小文字で示した。ColIb-P9において、相補性相互作用をすると考えられる7+3塩基からなる相補性塩基配列はドットで示した。 図3 a.in vitroで作製したキメラプラスミドpRC1、pRC2、pRC3の構造を模式的に示した。ColIb-P9の配列は白抜きの長方形で、R100の配列は影を付けた長方形で示した。縦の矢印はRNAの二次構造I、II、IIIの位置を示す。Pはプロモーターを示す。pRC1はR100のrepA1遺伝子の2番目のコドンをColIb-P9のrepZの5番目のコドンと接続して作製した。pRC2はR100のrepA6の7番目のコドンをColIb-P9のrepYの17番目のコドンと接続して作製した。pRC3はR100のG-528をColIb-P9のG-351に接続して作製した。b.ColIb-P9、R100、pRCC2、pRCC3の構造I上端部分の二次構造。ColIb-P9に由来する塩基は大文字で、R100に由来する塩基は小文字で示した。ColIb-P9において、相補性相互作用をすると考えられる7+3塩基からなる相補性塩基配列はドットで示した。 図表表2 キメラレプリコンの複製能とコピー数 / 表3 pRC1、R100のリーダーペプチドrepA6の翻訳が複製に与える影響 図表表2 キメラレプリコンの複製能とコピー数 / 表3 pRC1、R100のリーダーペプチドrepA6の翻訳が複製に与える影響 以上のことより、R100、ColIb-P9の複製領域は複製制御領域と複製構造領域とに機能的に区分でき、それぞれを組換えることにより、新しい複製システムが形成されることが実験的に証明できた。さらに複製可能なキメラプラスミドの形成は、複製制御に重要な役割を果たすRNA二次構造(複製タンパク質の翻訳開始シグナルを被う構造III、シュードノット)によって規定されていることを明らかにすることができた。 生物が多様性を獲得していく過程で制御遺伝子を組換えることが重要な戦略であることはこれまでに予想されてはいたが、塩基配列レベルで実例を見いだし、それをもとに、異なった生体システムを利用して天然に存在するものと同じ性質を保持したシステムを実際に作出したことは、おそらくこの研究が世界で最初であり、遺伝子システムの多様化の機構を解明する有効なモデルを提出することに成功した。 |