| 内容要旨 | | アミノアシルtRNA合成酵素(ARS)は遺伝暗号上で対応するアミノ酸に特異的なtRNAのみを厳密に認識してこれをアミノアシル化する.この認識は遺伝暗号の正しい解読に非常に重要な役割を果たしている.tRNAのアミノ酸特異性が、アイデンティティー決定因子と呼ばれる数残基のヌクレオチドによって決定されている例が近年数多く報告された.このため、ARSによるtRNAの認識機構の解析にあたっては残基レベルからの視点に重きが置かれてきた.しかし従来から、この認識が構造変化を伴う動的なものであることが示唆されており、またいくつかのtRNAではその三次構造がARSによる認識に重要であることが報告されている.つまり二次構造、三次構造を含めたtRNA分子全域にわたる解析を行う必要性がある.このような目的にはNMR法によるイミノプロトンシグナルの解析が有効な手段となりうるが、従来はシグナルの重なりなどから解析困難とされてきた.そこで本研究では安定同位体標識を利用してこれを克服し、認識機構を高次構造レベルから解析する事に成功した.対象としては大腸菌のイソロイシンおよびバリンの系に注目した.この二つの系は進化系統的に近いと考えられている.従来の解析では、ARSによるtRNAの認識は各アミノ酸の系ごとに異なると考えられていた.そこで系統的に近いアミノ酸の系での共通点、相違点を解析することで、認識機構に関する体系的な知見が得られるだけでなく、分子進化に関する情報をも得ることができると期待した. まず大腸菌のイソロイシンの系で解析を行なった.大腸菌tRNAIle1のNOESYスペクトルから、二次構造および三次構造塩基対のイミノプロトンシグナルをすべて帰属した.次に15N安定同位体均一標識された大腸菌tRNAIle1を調製して、2D1H-15N HMQCスペクトルを測定し、イミノプロトンシグナルの帰属に基づいて1H-15Nクロスピークの帰属を行った(図1a).そしてtRNAIle1と非標識の大腸菌イソロイシルtRNA合成酵素(IleRS)との複合体について同様に2D1H-15N HMQCスペクトルを測定して、tRNAIle1単独のHMQCスペクトルと比較した(図1b).その結果、多くのクロスピークに関しては、分子量の増加に伴うブロードニングが観測されただけであった.ところがいくつかのシグナルに関して、化学シフトの変化、強いブロードニング、さらにはクロスピークの消失といった現象が観測され、しかもこれらのクロスピークに対応するイミノプロトンはtRNA上の4領域に局在していた(図2).第一はアンチコドンステムである.C29-G41のイミノプロトンシグナルの化学シフトが変化し、C30-G40、C31-G39のシグナルが消失した(下線部の残基のイミノプロトンシグナルを観測している).イミノプロトンシグナルの消失は著しいブロードニングによるものと考えられるが、これはこの塩基対を含む領域に大きな構造変化が生じたことを示している.アンチコドンとC29-G41はtRNAIle1のアイデンティティー決定因子であり、アンチコドンアームはIleRSとの接触部位である(文献1).特にG41のカルボニル基がARSによる認識に強く関与していると考えられているが、これはメジャーグルーブ側に存在している(文献1).よってIleRSはこれを認識するためにアンチコドンステム領域に、複数塩基対の開裂といった大きな構造変化を引き起こしていると考えられる.第二の領域は領域はDステムである.C11-G24、U12-A23のイミノプロトンシグナルが強くブロードニングした.U12-A23がアイデンティティー決定因子であり、DステムはIleRSとの接触部位である(文献1).特にU12のマイナーグルーブ側のカルボニル基が認識に重要であると考えられている(文献1).よってIleRSはステム構造を保持したままこの領域と結合していると考えられる.第三の領域はアクセプターステム・Tステムの接合領域である.U6-A67のイミノプロトンシグナルの化学シフトが変化し、G7-C66のイミノプロトンシグナルは強くブロードニングした.また、G49-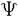 65、U5-G68のワブル塩基対に関しては、Gのイミノプロトンシグナルが消失したのに対し、Uのそれは影響が観測されていないことから、これらの塩基対はマイナーグルーブ側から歪みを受けていると考えられる.またC4-G69、A73がアイデンティティー決定因子であり、さらにC70、U72がIleRSとの接触部位であることから(文献1)、酵素がアクセプターステムの先端と結合してアイデンティティー決定因子を認識することで、アクセプターステムとTステムの接合領域に間接的で比較的小さな構造変化をもたらしていると考えられる.第四の領域はセントラルコア、バリアブルポケットである.G19-C56、G15-C48、G18- 65、U5-G68のワブル塩基対に関しては、Gのイミノプロトンシグナルが消失したのに対し、Uのそれは影響が観測されていないことから、これらの塩基対はマイナーグルーブ側から歪みを受けていると考えられる.またC4-G69、A73がアイデンティティー決定因子であり、さらにC70、U72がIleRSとの接触部位であることから(文献1)、酵素がアクセプターステムの先端と結合してアイデンティティー決定因子を認識することで、アクセプターステムとTステムの接合領域に間接的で比較的小さな構造変化をもたらしていると考えられる.第四の領域はセントラルコア、バリアブルポケットである.G19-C56、G15-C48、G18-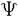 55のイミノプロトンシグナルが消失している.同じ三次構造塩基対U8-A14、G22-m7G46には影響が及んでいない.このことは前者の三塩基対が活性に必須でないのに対し、後者の二塩基対は活性に重要である(文献1)こととよく一致する.つまり特定の三次構造塩基対だけがIleRSの結合によって大きな影響を受け、それに伴いL字型三次構造そのものが変化する、そしてそのような構造変化が認識の特異性に必要であることが示唆された.以上より、IleRSはtRNAIle1のL字型構造の両端(アンチコドンと3’末端)だけでなく、ステムの中央の塩基対にあるアイデンティティー決定因子を認識するために、L字型構造の内側の広い範囲で接触し、それによってステム領域に直接的および間接的な局所的構造変化を引き起こし、さらにはグローバルな三次構造変化をも誘導するということが示された(図2).このグローバルな構造変化はX線結晶構造回折から明らかにされていた他の三種類のアミノ酸の系と比較して、非常に特徴的な現象であった.このようにtRNA分子全域における構造変化を同定し、認識機構との相関を明らかにしたことは画期的であり、他の系においても同様の手法が有効であることが示された. 55のイミノプロトンシグナルが消失している.同じ三次構造塩基対U8-A14、G22-m7G46には影響が及んでいない.このことは前者の三塩基対が活性に必須でないのに対し、後者の二塩基対は活性に重要である(文献1)こととよく一致する.つまり特定の三次構造塩基対だけがIleRSの結合によって大きな影響を受け、それに伴いL字型三次構造そのものが変化する、そしてそのような構造変化が認識の特異性に必要であることが示唆された.以上より、IleRSはtRNAIle1のL字型構造の両端(アンチコドンと3’末端)だけでなく、ステムの中央の塩基対にあるアイデンティティー決定因子を認識するために、L字型構造の内側の広い範囲で接触し、それによってステム領域に直接的および間接的な局所的構造変化を引き起こし、さらにはグローバルな三次構造変化をも誘導するということが示された(図2).このグローバルな構造変化はX線結晶構造回折から明らかにされていた他の三種類のアミノ酸の系と比較して、非常に特徴的な現象であった.このようにtRNA分子全域における構造変化を同定し、認識機構との相関を明らかにしたことは画期的であり、他の系においても同様の手法が有効であることが示された. ところで2D1H-15N HMQCスペクトル上に、アンチコドンに隣接する修飾ヌクレオシドt6A37NHのシグナルが観測された.このシグナルはIleRSの結合によって消失していた.tRNAIle1に関しては転写後修飾が活性に重要であることが当研究室で既に明らかにされていたが、具体的にどの修飾がARSによる認識に関与しているかは同定されていなかった.そこでこのt6A37を候補と考え、このt6Aの修飾のみを欠いた変異体をRNA分子整形法で調製した.この変異体のイソロイシン受容活性を測定すると、完全修飾tRNAIle1と比較してKm値は約3倍程度の上昇にとどまったのに対し、kcat値は約1/30に低下し、結果として約1/100に活性が低下した.完全未修飾tRNAIle1はKm値は約3倍程度の上昇に対して、kcat値は約1/150に低下して、結果として約1/500に活性が低下していた.このことから9残基の修飾ヌクレオシドの中でt6AがIleRSによる認識に最も大きな寄与をしていることが示された.このことは上記の認識機構のモデルとも一致する結果である. 次に同様の手法をバリンの系にも応用した.まず15N安定同位体均一標識された大腸菌tRNAVal1を調製して2D1H-15N HMQCスペクトルを測定し、イミノプロトンシグナルの帰属(文献2)に基づいて、1H-15Nクロスピークの帰属を行った(図3a).そしてtRNAVal1と非標識のバリルtRNA合成酵素(ValRS)との複合体について2D1H-15N HMQCスペクトルを測定して、tRNAVal1単独のHMQCスペクトルと比較し、シグナル解析を行った(図3b).その結果、ValRSの結合によって影響を受けたイミノプロトンを含む塩基対はイソロイシンの系と同様に、アンチコドンステム、Dステム、アクセプターステム・Tステム接合領域、バリアブルポケット、セントラルコアに集中していた(図4).次に大腸菌tRNAVal1とValRSとの相互作用を調べるために、アルキル化剤であるN-nitroso-N-ethylureaを用いたフットプリンティングの解析を行った.その結果、ValRSはtRNAVal1のアンチコドンからアンチコドンステムの3’側ストランドにかけてと、Dステム、およびアクセプターステムの先端と接触していることが明らかになった(図4).これはイソロイシンの系とほぼ同じである.また大腸菌tRNAVal1のアイデンティティー決定因子(文献3、4)の位置は大腸菌tRNAIle1の場合とほぼ同じである(図2、4).これらの解析において観測された類似性は、他のアミノ酸の系と比較して非常に際立っている.しかし各領域における構造変化には明らかな相違点も観測されている.例えばアンチコドンステムにおいては、tRNAIle1ではループ側3塩基対が影響を受けているのに対し、tRNAVal1では中央3塩基対が影響を受けている(図4).またtRNAIle1では主にバリアブルポケットに強い影響が観測されたのに対し、tRNAVal1ではバリアブルポケットとセントラルコア両域に影響が及んでいた(図4).よって大腸菌のバリンとイソロイシンの系では、ARSによるtRNAの認識機構は基本的な枠組みは共通であるものの、細部において両者を厳密に識別するためのメカニズムが用意されていると考えられる.  図表図1(a)15N-tRNAIle1の1H-15N2D HMQCスペクトル,クロスピークにシグナルの帰属を記した.(b)15N-tRNAIle1とIleRSとの複合体の1H-15N2D HMQCスペクトル。(a)に対応するピークが観測された場合にはその帰属を示した.下線を引いた残基はイミノプロトンシグナルの化学シフトがIleRSの結合によって0.03ppm以上変化したものを示した.枠で囲んだ残基は観測された他の残基よりもブロードニングが強かったものを示した.アステリスクは何らかのコンタミネーションによると考えられる. / 図2IleRSによるtRNAIle1の認識機構のまとめ.枠がIleRSの結合によって影響を受けたイミノプロトンシグナルを含む塩基対.■がシグナルが消失した塩基対を、 図表図1(a)15N-tRNAIle1の1H-15N2D HMQCスペクトル,クロスピークにシグナルの帰属を記した.(b)15N-tRNAIle1とIleRSとの複合体の1H-15N2D HMQCスペクトル。(a)に対応するピークが観測された場合にはその帰属を示した.下線を引いた残基はイミノプロトンシグナルの化学シフトがIleRSの結合によって0.03ppm以上変化したものを示した.枠で囲んだ残基は観測された他の残基よりもブロードニングが強かったものを示した.アステリスクは何らかのコンタミネーションによると考えられる. / 図2IleRSによるtRNAIle1の認識機構のまとめ.枠がIleRSの結合によって影響を受けたイミノプロトンシグナルを含む塩基対.■がシグナルが消失した塩基対を、 がシグナルの化学シフト値が変化した塩基対を、 がシグナルの化学シフト値が変化した塩基対を、 がシグナルのブロードニングが強かった塩基対を表す.ワブル塩基対は がシグナルのブロードニングが強かった塩基対を表す.ワブル塩基対は で示した.ワブル塩基対にはグアニン残基のシグナルは消失したのに対し、ウリジン残基のシグナルには影響が及んでいなかった.太線がフットプリンティング解析による、IleRSとの接触部位を表す.丸印がアイデンティティー決定因子を表す.フットプリンティングおよびt6A以外のアイデンティティー決定因子については文献1に従った. で示した.ワブル塩基対にはグアニン残基のシグナルは消失したのに対し、ウリジン残基のシグナルには影響が及んでいなかった.太線がフットプリンティング解析による、IleRSとの接触部位を表す.丸印がアイデンティティー決定因子を表す.フットプリンティングおよびt6A以外のアイデンティティー決定因子については文献1に従った. 図表図3(a)15N-tRNAVal1の1H-15N2D HMQCスペクトル.クロスピークにシグナルの帰属を記した。(b)15N-tRNAVal1とValRSとの複合体の1H-15N2D HMQCスペクトル.(a)に対応するピークが観測された場合にはその帰属を示した.枠で囲んだ残基はイミノプロトンシグナルの化学シフトがValRSの結合によって0.03ppm以上変化したものを示した.下線を引いた残基は観測された他の残基よりもブロードニングが強かったものを示した. / 図4 ValRSによるtRNAVal1の認識機構のまとめ.枠がValRSの結合によって影響を受けたイミノプロトンシグナルを含む塩基対.■がシグナルが消失した塩基対を、 図表図3(a)15N-tRNAVal1の1H-15N2D HMQCスペクトル.クロスピークにシグナルの帰属を記した。(b)15N-tRNAVal1とValRSとの複合体の1H-15N2D HMQCスペクトル.(a)に対応するピークが観測された場合にはその帰属を示した.枠で囲んだ残基はイミノプロトンシグナルの化学シフトがValRSの結合によって0.03ppm以上変化したものを示した.下線を引いた残基は観測された他の残基よりもブロードニングが強かったものを示した. / 図4 ValRSによるtRNAVal1の認識機構のまとめ.枠がValRSの結合によって影響を受けたイミノプロトンシグナルを含む塩基対.■がシグナルが消失した塩基対を、 がプロトンシグナルの化学シフト値が0.03ppm以上変化した塩基対を、 がプロトンシグナルの化学シフト値が0.03ppm以上変化した塩基対を、 がプロトンシグナルの化学シフト値が0.1ppm以上変化した塩基対を、 がプロトンシグナルの化学シフト値が0.1ppm以上変化した塩基対を、 がシグナルのブロードニングが強かった塩基対を表す.ワブル塩基対は がシグナルのブロードニングが強かった塩基対を表す.ワブル塩基対は で示した.ワブル塩基対にはグアニン残基のシグナルは消失し、ウリジン残基のシグナルの化学シフト値は変化した.太線がフットプリンティングの解析による、ValRSとの接触部位を表す.丸印がアイデンティティー決定因子(文献3、4)を表す. で示した.ワブル塩基対にはグアニン残基のシグナルは消失し、ウリジン残基のシグナルの化学シフト値は変化した.太線がフットプリンティングの解析による、ValRSとの接触部位を表す.丸印がアイデンティティー決定因子(文献3、4)を表す. 従来は、すべてのARSはそれぞれ大きく異なる認識機構を持つと推定されてきた.しかしながら、本研究の結果はこのような認識機構の差違が、tRNA/ARSの進化、分化とともに段階的に生じたことを示唆している.今後系統的に近いアミノ酸の系の間の比較を積み重ねることで、分子進化に関する重要な知見が得られることが期待される. 文献1 Nureki,O.,Niimi,T.,Muramatsu,T.,Kanno,H.,Kohno,T.,Florentz,C.,Giege,R.&Yokoyama,S.(1994)J.Mol.Biol.236,710-724文献2 Hare,D,R.,Ribeiro,N.S.,Wemmer,D.E.&Reid,B.R.(1985)Biochemistry24,4300-4306文献3 Tamura,T.,Himeno,H.,Asahara,H.,Hasegawa,T.&Shimizu,M.(1991a)Nucleic Acids Res.177,619-623文献4 Chu,W.-C.,Feiz,V.,Derrick,W.&Horowitz,J.(1992a)J.Mol.Biol.227,1164-1172 |
| 審査要旨 | | アミノアシルtRNA合成酵素(ARS)は遺伝暗号上で対応するアミノ酸に特異的なtRNAのみを厳密に認識してこれをアミノアシル化することで、遺伝暗号の正しい解読に重要な役割を果たしている.tRNAのアミノ酸特異性がアイデンティティー決定因子と呼ばれる数残基のヌクレオチドによって決定されている例が近年数多く報告されたため、ARSによるtRNAの認識機構解析にあたっては残基レベルからの視点に重きが置かれてきた. 本論文は、NMR法によってこの認識機構を高次構造レベルから解析し、ARSによる認識におけるtRNAの構造変化を同定することを目的としている.従来は、分子量の巨大さからNMR法をこの系に応用するのは困難であると考えられていたが、本研究では15N安定同位体均一標識をtRNA分子に導入することでこの問題点の克服を試みている.対象としては、大腸菌のイソロイシンおよびバリンの系に着目している.この二つの系は進化系統的に近縁であると考えられている.この二系における認識機構の共通点、相違点を解析することで、ARS/tRNAの分子進化に関する知見を得ることをも目的としている. 第一章では大腸菌イソロイシンの系に注目している.まず15N標識tRNAIle1を調製してイミノプロトンシグナルをすべて帰属し、次に非標識イソロイシルtRNA合成酵素(IleRS)を加えることで生じたスペクトルの変化を観測することにより、IleRSの結合によって誘導された構造変化を解析している.その結果、アンチコドンステムに塩基対開裂と考えられる強い構造変化が生じるほか、Dステム、アクセプターステムとTステム接合部位にも弱い構造変化が引き起こされることを明らかにしている.さらにTループ・Dループ会合が緩み、L字型三次構造に歪みが生じることをも示している.またNMR解析に基づいて、IleRSの結合によって影響を受けた修飾ヌクレオシドt6Aを検出し、RNA分子整形法によってt6Aの修飾を欠いた変異体を調製することで、このアンチコドンに隣接するt6Aが正及び負のアイデンティティー決定因子であることを明らかにしている.これらの結果と、既に報告されていた、変異体解析およびフットプリンティング解析の結果と総合することで、IleRSがtRNAIle1分子全体に散在するアイデンティティー決定因子を認識することによってtRNA分子上に、直接的、間接的な構造変化を誘導することを示した.以上のようにtRNA分子全域における構造変化を同定し、ARSによる認識との相関を詳細に明らかにしたことは画期的であり、このような手法は他のアミノ酸の系への応用性も高いと考えられる. 第二章では大腸菌のバリンの系に着目している.tRNAVal1についてフットプリンティング解析および1H-15N NMR解析を行なって、バリルtRNA合成酵素(ValRS)との接触部位および、ValRSの結合によって誘導される構造変化を同定し、さらに既に報告されている変異体解析の結果と総合することで、ValRSによるtRNAVal1の認識機構を明らかにしている.その結果、アイデンティティー決定因子の位置、ARSとの接触部位、ARSの結合によって生じる構造変化において、イソロイシンの系との相同性が高いことから、この二つの系においてはARSによるtRNAの認識機構の基本的な枠組みが共通であることを示している.また、それぞれのtRNAのアンチコドンアームに存在しているアイデンティティー決定因子が、この二つの系の識別に寄与していることをも示している. 第三章では、イソロイシン、バリンの系での解析結果と、他のアミノ酸の系において得られている結果との比較を行なっている.従来は、ARSによるtRNAの認識機構は各アミノ酸の系ごとに大きく異なっていると考えられていた.しかし本論文での研究は、そのような認識機構の差違がtRNA/ARSの進化、分化とともに段階的に生じたことを示している. 以上の研究においてtRNA,ARSの調製とNMR解析、フットプリンティング解析、修飾変異体の調製とその活性測定などすべて論文提出者が主体となって行なったものであり、審査委員会は本論文提出者が博士(理学)の学位を受ける資格があるものと判定した.なお本論文は、東京大学の横山茂之教授、渡辺公綱教授、武藤裕博士、河金剛太博士、宮澤辰雄東京大学名誉教授、東京工業大学の西川一八助教授、横川隆志博士、横浜国立大学の高柳正明氏、野口智明氏、三菱化成生命研究所の河野俊之博士、理化学研究所の濡木理博士、藤田保健衛生大学の林宣宏博士との共同研究であるが、論文提出者が主体となって分析および検証を行なったもので、論文提出者の寄与が十分であると判断する. |