| 内容要旨 | | 1984年,SchwartsとCantorらによって,パン酵母の各染色体の完全な長さを保持したDNA分子を単離する技術が開発された.染色体のDNAの調製はアガロース中で行なわれ,パルスフィールド電気泳動装置によって分画された.パルスフィールド電気泳動装置は数Mb(百万塩基)までの巨大DNAを分画するのに用いられる装置で,これによって,パン酵母の染色体が分離同定された.この巨大DNAを単離し分画するという新しい技術により,ひとつの遺伝子(の一部)から,染色体(の一部)にまで,解析のレベルが飛躍的に拡大された.植物において,巨大DNAの調製法を確立しパルスフィールド電気泳動法を用いて解析した例は,動物や下等真核生物に比較してはるかに少ない. そこで,本研究では,イネにおいて巨大DNAの調製法を開発し,イネ核ゲノムのDNA構造についてパルスフィールド電気泳動法を用いた解析を試み,以下の研究を行った. イネの核由来の巨大DNAの調製報の開発とパルスフィールド電気泳動法による解析 本研究では,初めて,イネの核由来の巨大DNAの調製法を確立した.すでに,トマトなど他の植物で巨大DNAの調製法が確立されていたが,プロトプラストを経由して調製する方法が多く用いられていた.これに対して,本研究では,胚芽から単離した核を用いて巨大DNAを調製した.プロトプラストを用いずに単離核を用いた理由は,イネでは,培養細胞以外の組織から得たプロトプロラストから,巨大DNAを得ることが難しいからである.また,胚芽を用いたのは,単離核調製時に用いる緩衝液に,核膜安定剤として,塩化マグネシウムの代わりにスペルミジンが用いられているので,エチレンジアミン四酢酸を加えることができ,デオキシリボヌクレアーゼ活性を抑えることが可能であったからである.胚芽から単離した核を,定法に従ってアガロースに包埋し,プロテアーゼと界面活性剤で処理して,核の巨大DNAをアガロース片として調製した.調製された核の巨大DNAをそのままパルスフィールド電気泳動したところ,5.7Mb以上の大きさを保持していることが分かった. 次に,定法に従って,アガロース片中で制限酵素処理を行ない,パルスフィールド電気泳動を行った.その結果,イネでは,平均200kb以上に切断する制限酵素がほとんど無いことが分かった.トマトやコムギなど,他の植物で,平均1Mb以上の制限酵素切断断片を生成することが,他の研究で明らかになっている,8塩基認識制限酵素,NotIを用いても,200kb以上の制限酵素切断断片は極めて少なく,大部分が50-100kbほどの大きさにとどまっていた.他の研究によって,イネでは核ゲノムDNAのメチル化が少ないことが分かっている.従って,この結果は,メチル化が少ないことを反映したものと考えられた. 次に,コピー数の少ない遺伝子である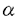 -アミラーゼ遺伝子RAmy1Aの検出を試みた.低塩濃度エレクトロブロット法によって転写を行ない,32P標識したプローブを用いてイメージングプレート法で検出することにより,当該遺伝子を検出することに成功した.さらに,RAmy1A遺伝子座の部分的な制限地図を作成することができ,パルスフィールド電気泳動法を用いた制限地図作成が可能であることが示された. -アミラーゼ遺伝子RAmy1Aの検出を試みた.低塩濃度エレクトロブロット法によって転写を行ない,32P標識したプローブを用いてイメージングプレート法で検出することにより,当該遺伝子を検出することに成功した.さらに,RAmy1A遺伝子座の部分的な制限地図を作成することができ,パルスフィールド電気泳動法を用いた制限地図作成が可能であることが示された. パルスフィールド電気泳動法を用いた多型性の検出 胚芽から核由来の巨大DNAを調製する方法は,胚芽を多量の種子から単離することが必要なため,種子の入手が困難な場合に,核の巨大DNAを調製するのは難しく,また,幾つかの試料植物について,少量ずつ核巨大DNAを調製することも困難であった.そこで,緑葉を材料として核巨大DNAを調製する方法を新たに開発した. 核由来の巨大DNAの調製に際しては,単離核を調製してそれを用いることを検討した.定法では,緑葉から核を単離する際には,塩化マグネシウムを含む緩衝液を用いる.しかし,塩化マグネシウムはデオキシリボヌクレアーゼを賦活化し,DNA切断を引き起こす.そこで,胚芽から核を単離する際に用いられる緩衝液と同様に,塩化マグネシウムの代わりにスペルミジンを用い,エチレンジアミン四酢酸を緩衝液に添加した.さらに,エチジウムブロミドも添加した.この緩衝液を用いて緑葉から単離核を調製し,定法に従って,巨大DNAを調製してパルスフィールド電気泳動を行ったところ,1.1Mb以上の巨大DNAが調製されているのが確認された. この方法を用いて,ジャポニカ種9品種,インディカ種1品種,及び野生稲Oryza punctataの1系統の,計11品種・系統のイネについて,巨大DNAを調製し,制限酵素SmaIで切断してパルスフィールド電気泳動を行った.以前より他の研究で,制限酵素切断した哺乳類の巨大DNAをパルスフィールド電気泳動すると,微細な明暗のバンドパターンが一様なバックグランド上に観察されることが知られていた.このようなパターンがこの実験でも観察された.これを写真撮影し,そのネガフィルムについてデンシトメーターで走査すると,各バンドがデンシトグラム上のピークとして同定された.各品種・系統にわたって,このようなデンシトグラムを得たところ,各ピークの存否や形状に多型性が観察されることが判明した.遺伝的に近縁であると考えられる3品種,「短銀坊主」・「早生銀坊主」・「銀坊主」では,そのデンシトグラムが他の品種に比してよく似ていた.また,電気泳動の条件を変更して同様な実験を行ったところ,これらの3品種の近似性は保存されていた.また,同じく近縁な2品種,「フジミノリ」と「レイメイ」の間でも,デンシトグラムに近似性が認められた.実験に供したジャポニカ種に属する品種の間では,共通なピークが幾つか観察されたのに対し,インディカ種である「ハバタキ」や,野生稲Oryza punctataのW1515系統との間に,共通したピークはほとんど見られなかった.以上の実験結果によると,核の巨大DNAを制限酵素SmaIで処理して,パルスフィールド電気泳動した後に,ゲルを染色して観察される明暗の微細なバンドパターンに,遺伝的な近縁性を反映した多型性が検出されたものと考えられる. リボソームRNA遺伝子座の構造解析 次に,リボソームRNA遺伝子座の構造解析を試みた.この遺伝子座では,数百回以上連続してリボソームRNA遺伝子配列が繰り返していることが知られている.この遺伝子座をパルスフィールド電気泳動法によって解析するに当たって,材料とした品種「日本晴」について,すでにクローン化されていた品種「マンゲツモチ」のリボソームRNA遺伝子クローンをプローブとして,リボソームRNAの遺伝子のクローン化を行った.この「日本晴」のクローンには,HindIII,PstI,SplIの三種類の制限酵素の切断部位が存在しなかった.そこで,イネ核巨大DNAをこれらの制限酵素で切断しパルスフィールド電気泳動法で分画して,リボソームRNA遺伝子をプローブとしてハイブリダイゼーションを行ったところ,1.1Mb以上の領域に当該遺伝子を含む巨大な制限酵素切断断片が存在することが確認された.従って,リボソームRNA遺伝子は,少なくともこれらの制限酵素切断部位が存在しないことに関して,1.1Mbすなわち130以上の繰り返し単位にわたって(8kb/1繰り返し単位)均一に繰り返していることが示唆された.また,当該遺伝子クローンによると,切断部位のメチル化で切断できなくなる制限酵素である,SalI,MluI,NotIの各制限酵素の切断部位が複数存在している.これらの制限酵素を用いて,同様な実験を行ったところ,サザン解析において,色々な制限酵素切断断片にリボソームRNA遺伝子が検出された.これにより,イネの胚芽において,リボソームRNA遺伝子繰り返し単位の当該制限酵素切断部位が,メチル化されているものと,メチル化されていないものが存在し,その二種類の繰り返し単位が,不均一に存在していることが示唆された. 次に,リボソームRNA遺伝子配列の繰り返しの末端部のクローン化を試みた.イネ核巨大DNAをリボソームRNA遺伝子を切断しない制限酵素HindIIIを用いて切断して,パルスフィールド電気泳動法によって数百kb以下の断片を除去して,リボソームRNA遺伝子座を含む巨大なHindIII切断断片を濃縮した.これをリボソームRNA遺伝子を1箇所切断するEcoRIで処理した.このとき,リボソームRNA遺伝子座末端部に相当する断片は,HindIII切断端(非リボソームRNA遺伝子側)を片方に,EcoRI切断端(リボソームRNA遺伝子側)をもう一方の端にもつはずである.そこで,この断片をHindIII切断端をもつ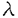 BlueMidベクターDNAの左腕と,EcoRI切断端をもつ BlueMidベクターDNAの左腕と,EcoRI切断端をもつ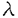 BlueMidベクターDNAの右腕につないで,選択的にクローン化し,リボソームRNA遺伝子をプローブとして探索したところ,3つのクローンが得られた.No.1とNo.2のクローンは(転写方向に対して)上流側末端のクローンに相当し,25SリボソームRNA遺伝子の途中からリボソームRNA遺伝子の繰り返しが始まっていた.また,No.3のクローンは下流側末端のクローンに相当し,(遺伝子を直接コードしない)スペーサー領域の終端部で,リボソームRNA遺伝子領域が終了していた.これらのクローンを,制限酵素切断断片長多型により地図上に位置付けることができれば,遺伝子が,染色体基部から末端部へ転写されるのか,あるいはその逆かが明らかになるであろう. BlueMidベクターDNAの右腕につないで,選択的にクローン化し,リボソームRNA遺伝子をプローブとして探索したところ,3つのクローンが得られた.No.1とNo.2のクローンは(転写方向に対して)上流側末端のクローンに相当し,25SリボソームRNA遺伝子の途中からリボソームRNA遺伝子の繰り返しが始まっていた.また,No.3のクローンは下流側末端のクローンに相当し,(遺伝子を直接コードしない)スペーサー領域の終端部で,リボソームRNA遺伝子領域が終了していた.これらのクローンを,制限酵素切断断片長多型により地図上に位置付けることができれば,遺伝子が,染色体基部から末端部へ転写されるのか,あるいはその逆かが明らかになるであろう. |
| 審査要旨 | | 植物の巨大DNAの調製法を確立し,パルスフィールド電気泳動法を用いて解析した列は,動物や下等真核生物に比較してはるかに少ない。本研究では,イネの巨大DNAの調製法を開発し,イネ核ゲノムのDNA構造についてパルスフィールド電気泳動法を用いた解析を試みた。 1.イネの核由来巨大DNAの調製法の開発とパルスフィールド電気泳動法による解析 イネの核由来の巨大DNAの調製法を胚芽から単離した核を用いて確立した。アガロース片中に調製された巨大DNAをそのままパルスフィールド電気泳動したところ,5.7Mb以上の大きさを保持していることが示された。 次に,アガロース片中で制限酵素処理を行ない,パルスフィールド電気泳動を行った。その結果,イネでは平均200kb以上に切断する制限酵素がほとんど無いことが明らかになった。トマトやコムギなど他の植物で,平均1Mb以上の制限酵素切断断片を生成することが,他の研究で示されている,8塩基認識制限酵素,NotIを用いても,200kb以上の制限酵素切断断片は極めて少なく,大部分が50〜100kbほどの大きさにとどまっていた。この結果は,メチル化が少ないことを反映したものと考えられた。 また,コピー数の少ない遺伝子である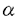 -アミラーゼ遺伝子RAmy 1Aの検出を行ない,RAmy 1A遺伝子座の部分的な制限地図を作成した。 -アミラーゼ遺伝子RAmy 1Aの検出を行ない,RAmy 1A遺伝子座の部分的な制限地図を作成した。 2.パルスフィールド電気泳動法を用いた多型性の検出 胚芽から核由来の巨大DNAを調製する方法は,胚芽を多量の種子から単離することが必要なため,種子の入手が困難な場合に,核の巨大DNAを調製するのは困難であった。そこで,緑葉を材料として核巨大DNAを調製する方法を新たに開発した。 この方法を用いて,ジャポニカ亜種9品種,インディカ亜種1品種,及び野生種Oryza punctataの1系統の,計11品種・系統のイネについて,巨大DNAを調製し,制限酵素SmaIで切断してパルスフィールド電気泳動を行ったところ,各レーンに微細な明暗のバンドパターンが観察された。これを写真撮影し,そのネガフィルムをデンシトメーターで走査し,各バンドの存否や形状に多型性が観察されることを明らかにした。遺伝的に近縁である品種,「短銀坊主」・「早生銀坊主」・「銀坊主」では,そのデンシトグラムがよく似ていた。ジャポニカ亜種に属する品種の間では,共通なバンドが幾つか観察されたのに対し,インディカ亜種である「ハバタキ」や,野生稲Oryza punctataのW1515系統との間に,共通したバンドはほとんど観察されなかった。以上の実験結果から,これらの微細なバンドパターンを遺伝的な近縁性を反映した多型性と考えた。 3.リボソームRNA遺伝子座の構造解析 リボソームRNA遺伝子はMb単位にわたって繰返し構造をしていることが,報告されている。そこでこの構造の解析を試みた。遺伝子内に切断部位のない制限酵素で,イネ核巨大DNAを切断しパルスフィールド電気泳動法で分画して,リボソームRNA遺伝子をプローブとしてハイブリダイゼーションを行ったところ,1.1Mb以上の領域に当該遺伝子を含む巨大な制限酵素切断断片が存在することが確認された。このことにより,130コピー以上にわたって,リボソームRNA遺伝子が連続して繰り返していることが示された。切断部位のメチル化で切断出来なくなる制限酵素を用いて,同様な実験を行ったところ,色々な制限酵素切断断片にリボソームRNA遺伝子が検出された。これにより,リボソームRNA遺伝子繰り返し単位の当該制限酵素切断部位が,メチル化されているものと,されていないものが存在し,その二種類の繰り返し単位が,不均一に存在していることが示唆された。 次に,リボソームRNA遺伝子の繰り返しの末端部のクローン化を試み,3つのクローンが得られた。これらのクローンを,制限酵素切断断片長多型により地図上に位置付けることが,今後の課題である。 以上要約するに,イネの核から巨大DNAを抽出し,パルスフィールド電気泳動で分画する技術を確立すると共に,それを用いて,イネ品種間の多型の検出やリボソームRNAの遺伝子構造を解明した。 よって、審査員一同は本論文が博士(農学)の学位を授与するに値するものと認めた。 |