| 1.序文 真核生物の細胞周期は外界からの情報によって正または負に制御されている。外界からの情報は様々な生理活性物質として、細胞の表面の受容体を通じて細胞内の種々の情報伝達経路を活性化し、最終的にDNA複製、転写、細胞分裂などを机御する。細胞外シグナルによる細胞周期の制御機構には未解決の重要な問題が数多く存在するが、そのなかのひとつであるDNA複製開始機構はG1-S移行で最も重要な過程であり、増殖因子による外界からの情報伝達の最終的な標的の一つである。 原核生物のDNA複製及び複製開始機構については大腸菌をモデルとして、非常に詳細な研究が行われている。真核生物のDNA複製の生化学的機構は基本的には原核生物の場合と類似していることが示されつつあるが、その制御機構についてはまだ解明が進んでいない。原核生物と真核生物のいずれの場合もDNA複製制御の重要な調節点はDNA複製開始にあると考えられる。原核生物では、培地の栄養源に応答してDNA複製開始の頻度が調節されている。しかし、原核生物ではDNA複製と細胞分裂は同時に進行しうるのに対し、真核生物では細胞分裂はDNA複製の完了に依存して開始し、複製開始は細胞分裂の完了に依存する。従って真核生物では原核生物には存在しない付加的な制御機構が存在することが示唆される。 酵母は遺伝子破壊、相補性テスト、ゲノムヘの変異の導入、安定なエピゾームベクターの存在、世代時間が短い、といった点で真核生物の有益なモデル生物であり、DNA複製開始機構のように真核生物全般に保存されていることが期待される現象を解析するために適している。さらに、様々な細胞周期変異株cdc(cell division cycle)が得られており、遺伝学的に詳細に解析されている。特に出芽酵母では50を越えるcdc変異(温度感受性)株が存在し、非許容温度下で、細胞周期の様々な位置で停止する変異が存在する。このcdc温度感受性変異の中で遺伝学的に最もDNA複製開始に近い時期に位置しているのがcdc7である。 cdc7変異は、細胞周期の重要な制御分子として知られているcdc28変異の下流に位置している。その形態はダンベル型で、このことは細胞質や紡錘体の分裂は進行しているにもかかわらず、DNA複製は開始していないことを示している。また、cdc7を非許容温度から許容温度に戻した場合に新たにタンパク質を合成することなくDNA複製を開始することができる。これらのことから、cdc7はDNA複製の直前で働いていることが予想される。このcdc7変異を相補する遺伝子としてクローニングされたCDC7遺伝子は58kDaからなるセリン-スレオニン型タンパク質キナーゼをコードしている。このキナーゼの発現量は細胞周期を通してほぼ一定であるが、その活性はG1-S移行期で最大に上昇する。キナーゼ活性の制御はcdc7の多コピー相補遺伝子としてクローニングされたDbf4が担っていると考えられる。Dbf4はG1-S移行期でその転写量が最大に達する。また、Cdc7とDbf4が相互作用することがtwo hybrid systemを用いて示されている。最近、Dbf4が出芽酵母の複製起点であるARS(autonomously replicating sequence)と結合することがone hybrid systemによって示されており、このことからも、Cdc7-Dbf4複合体がDNA複製開始に直接的に働いている可能性が強く示唆された。 このように、CDC7が出芽酵母のDNA複製開始制御に関与しているといういくつかの証拠は存在するがその機能や生理的な基質が未解明であるという問題は残されている。さらに、現在までのところCDC7遺伝子は出芽酵母(Saccharomyces cerevisiae)でしか発見されておらず、複製起点の構造が出芽酵母とは異なることが示唆されている高等動物においてもCDC7による制御機構が保存されているかどうかは不明である。そこで、本研究では、出芽酵母よりも進化的にもかなり離れており、複製起点の構造も高等動物と出芽酵母の中間的な性質を持ち、モデル生物としても有効である分裂酵母(Schizosaccharomyces pombe)でCDC7の相同遺伝子を検索し、その機能解析を通じて真核生物全般に共通な複製開始制御機構の一端を明らかにすることを目的とした。 2.結果 分裂酵母のCDC7相同遺伝子のcDNA断片をプローブとしてゲノムDNA断片のクローニングを行い、7.4kbpのEcoRIゲノム断片を取得した。cDNA全長およびゲノムDNA中の約3.6kbpの塩基配列を決定したところ、この遺伝子には、2つのイントロンがあり、507アミノ酸からなるセリン-スレオニン型タンパク質キナーゼがコードされていた。予想される分子量は58370Daで出芽酵母のCdc7とアミノ酸レベルで46%の相同性、65%の類似性を持っており、キナーゼドメインに限って調べると58%の相同性、76%の類似性を示した。さらに、CDC7遺伝子に特徴的な3つのキナーゼ挿入配列が同様な位置に存在し、C末端に酸性アミノ酸に富んだ配列を有していた。このことから、この遺伝子をhsk1+と名付けた(homologue of CDC7[seven]kinase)(図参照)。サザンハイブリダイゼーションの結果よりhsk1+遺伝子は全ゲノム中に1コピー存在することが示された。 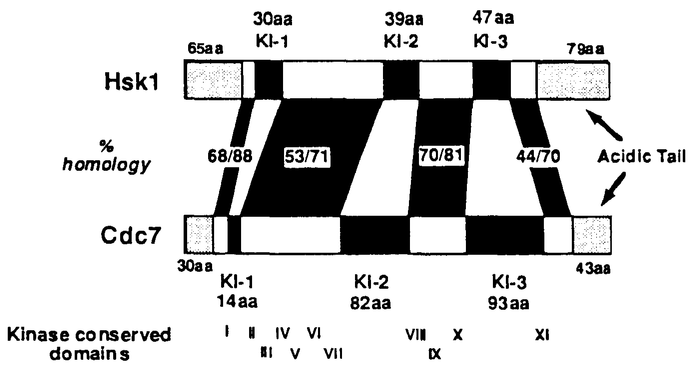 図表 図表 2倍体のゲノム上のhsk1+遺伝子をura4+遺伝子を用いて遺伝子破壊してその株を四分子解析したところ2:2で致死性を示したことから、この遺伝子は必須遺伝子であることが示された。この四分子解析の生育しなかった細胞は、約70%の細胞が2つの細胞がつながった形で生育が停止しており、残りの30%は数回細胞分裂したところで生育が停止していた。いずれの場合も細胞が異常に伸長しているものはほとんど観察されなかった。この形態は分裂酵母のDNA polymerase  の遺伝子破壊株の形態と類似していた。さらに、この遺伝子破壊株を胞子から発芽させてそのDNA含量をFlow cytometryを用いて解析し、核染色による形態観察を行ったところ、hsk1+遺伝子破壊株はDNA複製開始及び細胞分裂が異常であることが示された。これらの結果はHsklが、DNA複製開始および細胞分裂において重要な役割を果たしていることを示唆するものである。 の遺伝子破壊株の形態と類似していた。さらに、この遺伝子破壊株を胞子から発芽させてそのDNA含量をFlow cytometryを用いて解析し、核染色による形態観察を行ったところ、hsk1+遺伝子破壊株はDNA複製開始及び細胞分裂が異常であることが示された。これらの結果はHsklが、DNA複製開始および細胞分裂において重要な役割を果たしていることを示唆するものである。 hsk1+のcDNAを出芽酵母のGALプロモーターの下流に挿入して3種類の出芽酵母cdc7株に導入したが、いずれの場合も非許容温度下で生育することができず、また過剰発現しても生育に影響を及ぼさなかった。また、hsk1の遺伝子破壊株に出芽酵母のCDC7を導入した場合も致死性を回復することはできなかった。 3.考察と展望 分裂酵母から出芽酵母のCDC7キナーゼ相同遺伝子(hsk1+)のゲノムDNAをクローニングしてその一次構造を決定し、遺伝子破壊により必須遺伝子であることを示した。さらに遺伝子破壊株の形態からhsk1+がDNA複製開始および細胞分裂に関与していることが示唆された。 Hsk1とCdc7は互いに相補することができなかったが、これはDbf4が相互作用するとされているC末端側の1次構造が異なるためhsk1とDbf4、あるいはCdc7と分裂酵母Dbf4相同遺伝子が相互作用できないことが原因の一つと考えられる。 出芽酵母CDC7キナーゼの相同遺伝子が分裂酵母からクローニングされたことは以下の点で重要な意味を持つ。第1に、現在までSaccharomyces cerevisiae以外の生物でCDC7相同遺伝子は発見されておらず、本研究はCDC7相同遺伝子のクローニングとしては初めての報告である。分裂酵母は出芽酵母と進化的に離れており、この2種の酵母にCDC7キナーゼ相同遺伝子が存在したという事実は動物を含む他の真核生物に広く存在する可能性を強く示唆する。第2に、hsk1+がDNA複製開始および細胞分裂の進行に関与する点である。hsk1+はCDC7と同様に必須遺伝子であり細胞周期の進行に関与するということは、分裂酵母の細胞周期の解明の重要な糸口の1つとなると考えられる。 今後はさらにhsk1+の詳細な機能解析を行うとともにhsk1+遺伝子を用いてDBF4相同遺伝子を含むDNA複製開始制御機構に関与する他の遺伝子をクローニングし、それらの解析を行い、真核生物複製開始制御機構の理解に貢献したい。 |