一般に薬物は、標的となる生体高分子(以下受容体と呼ぶ)と強い安定な相生作用をすることによって、その薬理作用を発現する。受容体-リガンド間の認識に重要なのは、リガンドが特定の分子骨格を持つことではなく、2分子が十分に安定な複合体を形成することである。この安定化に寄与するのはvdW相互作用、水素結合、静電相互作用、疎水相互作用などである。従って、1つの受容体に対して多様な分子骨格をもつリガンドが、未知のものも含め多数存在するはずである。しかし、それらを見出すリード創製(発見)の論理的かつ効率的な方法はこれまで無く、膨大な化合物ライブラリを用いた実験的スクリーニングが唯一確実な手段であった。近年、生体物質の単離精製や構造決定の技術のめざましい発展により、生体高分子が次々に単離され、その機能や立体構造の解明が急速に進みつつある。これに伴い、受容体の立体構造情報を利用し、コンピュータを用いて論理的にリード創製を行う研究に関心が高まっている。そのアプローチとして、リードとなりうる構造を、既存化合物から探す三次元構造データベース検索法と、コンピュータに自動的に構築させる方法の2つが考えられる。後者は示唆される構造の多様性の点で優れており、前者は市販品や社内化合物など入手可能な化合物群を対象にすることで、ヒットした化合物を合成せずに入手し活性を評価できる点が魅力である。 データベース検索法については、既知活性化合物の情報から活性発現に必須と推定される官能基等(pharmacophore)の有無や位置関係を検索の条件(query)とする方法が近年多数開発されている。アルゴリズムが単純で、受容体の立体構造が未知であっても利用できるなど利点はあるが、pharmacophoreの条件はリガンドとなるための条件の1部でしかないため、ヒット化合物が実際に活性をもつ確率は高くない。受容体の立体構造が既知の場合には、そのリガンド結合部位への相互作用の強さが最も有効な検索queryであり、はるかに高い予測性をもって新規の活性化合物を見出すことができると考えられる。相互作用の強さは、最も安定な複合体を形成したときの構造において評価されなければならない。従って、この検索法を実現するためには、最安定複合体構造を確実に探索できるドッキング法がまず必要になる。 リガンド単独での構造(結晶中、溶液中、エネルギー安定構造etc.)におけるコンフォメーションと、受容体に結合したときの活性コンフォメーションとは一致する必然性がない。従って、ドッキングの際、コンフォメーションの自由度を考慮しなければ、正しい安定複合体構造が得られる保証はない。ドッキング法の開発にあたって最も難しい問題は、結合様式の自由度と化合物のコンフォメーションの自由度から生じる天文学的な数の可能性をいかに網羅するか、ということである。特に、コンフォメーションについては、その余りに膨大な可能性を扱いきれず、入力構造に固定してドッキングを行っているのが現状である。この困難な問題を解決する手法がないために、一般には、コンピュータグラフィックスを用いて対話的に求めた安定そうなドッキングモデルに対し、分子力学・動力学計算、分子軌道法などでエネルギー評価や最適化を行う手法がとられてきた。しかしこの方法では、コンフォメーションどころか結合様式に関する可能性さえ網羅することはできない。以上のような自由度の問題をうまく解決できる自動ドッキング法の開発は非常に困難であり、1個の化合物を対象とする場含ですら、世界的にも成功した例はない。 私は、受容体の立体構造が既知のときに、既存化合物から新しい生理活性を見出すことを目標に定め、まず、受容体-化合物間の相互作用を定量的に見積もりながら、結合様式、化合物コンフォメーションについての膨大な可能性を効率よく網羅できる高速な自動ドッキング法ADAMを開発した。次にその方法を応用して、三次元構造データベース中の数万-数十万分子から標的受容体のリガンドとなりうる化合物を検索する方法ADAM&EVEの開発を行った。 【1個の化合物を対象とした自動ドッキング法ADAM】 ADAM開発の基本方針を記す。1.分子間相互作用を重視。2.相互作用の指標はエネルギー値。3.結合様式と化合物のコンフォメーションの可能性を網羅。4.計算に用いるリガンド結合領域は予め指定するが、それ以外の先入観は一切入れない。5.自動的かつ高速であること。 ADAMの方法の流れを図1に示す。受容体側に、まずリガンド結合領域を指定する。そこに種々のポテンシャル値が格納された三次元格子点を発生させ、同時に、リガンドの水素結合性原子の存在が予測される位置にダミー原子をおく。化合物は水素結合性官能基を含む部分と含まない部分とに分けてドッキングを行う。化合物の水素結合性部分のコンフォメーションを系統的に変化させ、受容体のダミー原子と化合物の水素結合性原子の距離関係が一致するような候補コンフォマーを選んで最適化する。このとき全ての水素結合対の組み合わせを考慮することによって、可能な結合様式を網羅する。得られた候補コンフォマーを受容体にはめ込み、格子点情報を用いて水素結合部分の相互作用エネルギーの評価、構造最適化を行う。その後、非水素結合性部分の結合様式、コンフォメーションの探索を系統的に行い、全体のエネルギー評価、構造最適化を経て数個-数十個のドッキングモデルを得る。更に、水分子の存在と受容体構造のflexibilityまで考慮したエネルギー極小化を行って、順位付けされた安定複合体モデルを得る。  図1 自動ドッキシグ法ADAMの流れ 図1 自動ドッキシグ法ADAMの流れ ADAMを13個の酵素-リガンドの系に適用したところ、得られた最安定複合体モデルはどれも結合様式、リガンドのコンフォメーションともに結晶中の複合体構造を非常によく再現していた。 【三次元データベース検索法ADAM&EVE】 次に、ADAMを三次元データベース中の大量の化合物に次々と自動的に適用できるよう、アルゴリズムをmodifyした。高速化のためにパラメタのチューニングも行った。 ADAM&EVEの構成を図2に示す。検索対象とする低分子化合物群は、非水素原子の三次元座標と原子間の結合情報さえ持っていれば、どのような三次元構造データベースから得てもよいが、主に市販化合物、自社化合物や結晶構造のデータベースが用いられる。ADAMを行うために必要な以下のデータを自動的に追加して、ADAM&EVE仕様の三次元データベースを構築する。(1)水素原子の座標(2)各ヘテロ原子の水素結合タイプ(水素結合パターン推定のため)(3)各原子の原子電荷と力場原子タイプ(エネルギー算出、構造最適化のため)(4)結合回転に関する条件設定(コンフォメーションの自由度を扱うため)。modifyされたADAMのアルゴリズムを用いて、各化合物と受容体との自動ドッキングを次々と試みる。各化合物について得られた最安定モデルの相互作用エネルギー、水素結合数等を判定基準としてリガンド候補が選ばれ、モデルの三次元座標も出力される。更に検索終了後に、より厳しい条件で有望なリガンド候補を絞り込む。 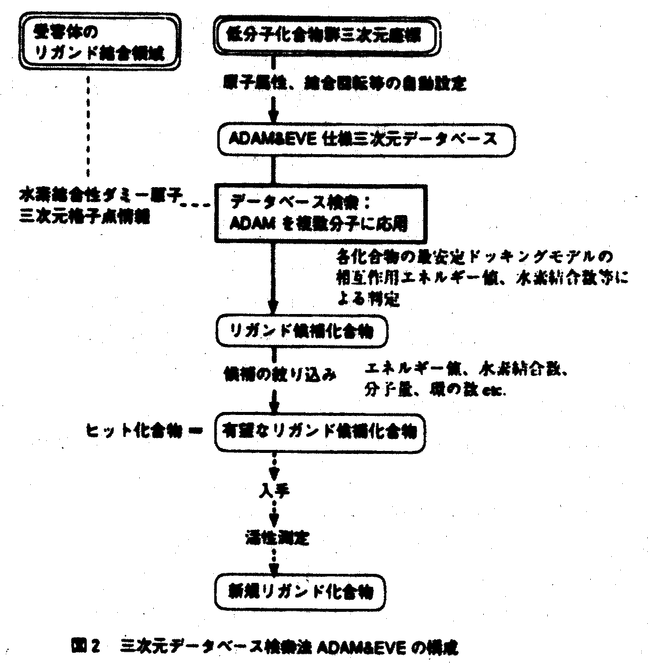 図2 三次元データベース検索法ADAM&EVEの構成 図2 三次元データベース検索法ADAM&EVEの構成 本方法の有効性を検証するために、HIV-1 proteaseを対象としたデータベース検索を行ってみた。検索対象とした低分子化合物は、約11万件の市販化合物のデータを含むACD-3D(MDL社)から得た。今回は6ヶ所以上の結合回転がある分子を除外した検索を行い、224個の多様な分子骨格をもつヒット化合物を得た。一部を図3に示す。視覚的判断も交えて少数の候補をピックアップし、現在までに人手できた12分子について、酵素に対する阻害活性の測定を行った。6分子には、分子量の大きいペプチド性阻害剤pepstatin Aには及ばないながらも、非ペプチド性阻害剤haloperidolより強い阻害活性が認められた。上位4化合物及び2つの既知阻害剤の50%阻害濃度(IC50)の測定結果を表1に示す。本来はここで、各化合物が基質と競合的な阻害様式であるかどうかを調べ、阻害物質定数Kiも測定すべきであるが、十分に精製された酵素がまだ人手できないため、現段階では実験を行っていない。そこで、各化合物のIC50の実験値と、既知阻害剤のKiの文献値から、仮にKi値を推定した(表1)。どの化合物も、Ki値は10-6Mのオーダーであることが予想され、医薬品のリードとして充分な活性であると考えられる。今後、適切な構造修飾を施したものを合成し、活性の増強を図りたい。  図3 ヒット化合物の例(標的酵素に対する最安定ドッキングモデル)・ヒットの条件:(1)相互作用エネルギー≦-8kcal/mol(分子量100あたり)(2)水素結合≧3(3)原子数≧30(4)環構造を合むetc. ・鳥カゴ状の表示はリガンド結合部位の形状を表す。#1〜4は表1の化合物と対応する。 図3 ヒット化合物の例(標的酵素に対する最安定ドッキングモデル)・ヒットの条件:(1)相互作用エネルギー≦-8kcal/mol(分子量100あたり)(2)水素結合≧3(3)原子数≧30(4)環構造を合むetc. ・鳥カゴ状の表示はリガンド結合部位の形状を表す。#1〜4は表1の化合物と対応する。 表1 入手した化合物の阻害活性 表1 入手した化合物の阻害活性 ジヒドロ葉酸還元酵素に対しても同様の検索を行ってみたところ、基質や既知阻害剤のアナログが多数ヒットし、新規リガンドも見出された。 【まとめと考察】 1.世界で初めて、結合様式と化合物コンフォメーションについての可能性をくまなく探索して最安定複合体モデルを構築できる自動ドッキング法の開発に成功した。 2.1の方法を利用し、受容体への相互作用の強さを検索queryとして、三次元データベースから新規リガンドの有望な候補を選び出す方法の開発に成功した。 3.HIV-1 proteaseの系で、新規な活性化合物が6個見出され、本検索法の高い予測性が実験的に証明された。 4.医薬開発におけるリード創製への有用性のみならず、本検索法は、立体構造情報さえ得られていれば、あらゆる生体高分子について、その機能を選択的に制御する人工リガンドを創製することが可能であり、それを通じて生体メカニズムを解明するための重要なツールとなることが期待される。 |