本論文は、カイコから単離された、昆虫で最初のインスリン族ペプチドであるボンビキシンについて、その受容体の解明を目的として実施された研究結果をまとめたもので、本文2部、緒言、総括および実験の部より構成されている。 第1部は、ボンビキシン受容体タンパク質の解析に関するものである。まず、6種のカイコ培養細胞系について、ボンビキシンに対する反応の有無を検討した。その結果、BM-N4系細胞が10-9M以上のボンビキシンに対して形態変化を伴う反応を示すことを見いだした。この発見は、これまでカイコにおける機能が明らかにされていないボンビキシンについて、ボンビキシンがカイコにおいて機能を有する可能性を細胞レベルにおいて示したものとして注目される。また、インスリンやリレキシンのようなインスリン族ペプチドにおいてはそのような形態変化は認められず、BM-N4細胞に、ボンビキシン受容体が発現している可能性の高いことが示された。 次にヨウ素ラベルしたボンビキシンを用いてBM-N4細胞に対する特異的結合量を測定した。スキャッチャード解析の結果からは、BM-N4細胞には、1細胞当たり約1万5千の受容体(解離定数、Kd=2.36±0.56nM)が発現していると推定された。さらに、化学架橋剤を用いてリガンドー受容体複合体を形成し、細胞を可溶化した後、還元および非還元条件下でSDS-PAGEを行ったところ、還元条件では約105kDa、非還元条件下では約300kDaの位置に、リガンドと特異的に結合したと思われるバンドが検出された。この結果から、ボンビキシン受容体はジスルフィド結合を介するサブユニット構造をとっていることが示唆された。また、抗ホスホチロシン抗体を用いたウエスタンプロッティングにより、ボンビキシンの刺激により速やかにリン酸化される約92kDaのバンドの存在を認めた。 インスリン受容体の サプユニット(約92kDa)は、インスリンの刺激により自己リン酸化されることが知られており、これらの結果を総合して、本論文の著者はボンビキシン受容体は、インスリン族ペプチドの受容体と類似の構造を有するものと推論した。 サプユニット(約92kDa)は、インスリンの刺激により自己リン酸化されることが知られており、これらの結果を総合して、本論文の著者はボンビキシン受容体は、インスリン族ペプチドの受容体と類似の構造を有するものと推論した。 第2部は、ボンビキシン受容体遺伝子のクローニングに関するものである。 本論文の著者は、ボンビキシン受容体がインスリン受容体と類似の構造を有することが推定された事実から、それらの間に相同性が存在することを仮定し、相同性に基づいたcDNAのクローニングを試みた。インスリン受容体間でよく保存されているチロシンキナーゼ領域について複数の混合プライマーを作成しRT-PCRを行ったところ、チロシンキナーゼ領域と相同性の高い配列を有するDNA配列が増幅された。この配列に対するantisense primerを用いてcDNAを合成しライブラリーを構築、スクリーニングを行ったが期待されるクローンは得られなかった。そこで、5’-RACE法、および、3’-RACE法を用いて、5’および3’側の非翻訳領域のクローンを得た。このクローンの塩基配列を決定し翻訳アミノ酸を解析したところ、図1に示すように、インスリン受容体の構造と極めて類似した構造を有することが示された。このクローンが真にボンビキシン受容体に対応するものであるか否かは、さらに検討を有するが、無脊椎動物インスリン族ペプチドに対する受容体は未だクローニングされておらず、ここにその有力な候補が得られたことは、高く評価されよう。 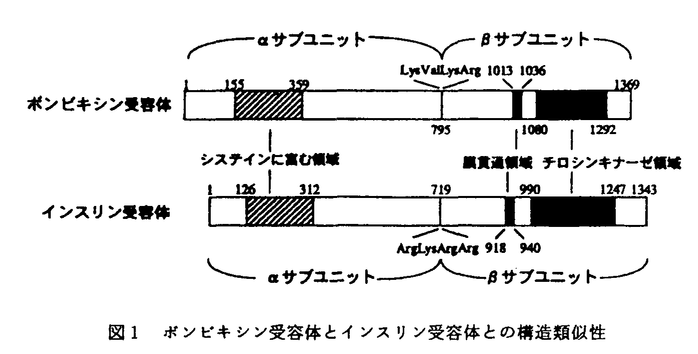 図1 ボンビキシン受容体とインスリン受容体との構造類似性 図1 ボンビキシン受容体とインスリン受容体との構造類似性 以上、本論文は昆虫インスリン族ペプチド、ボンビキシンの受容体タンパク質について解析を行うと共に、その遺伝子のクローニングを試みた結果について論じたもので、学術上貢献するところが少なくない。よって、審査員一同は、本論文が博士(農学)の学位請求論文として価値あるものと認めた次第である。 |