イネ(Oryza属)はいくつかのゲノムグループに分類されるが、中でもアジア栽培稲(Oryza sativa)、アフリカ栽培稲(O.glaberrima)を含むAAゲノムをもつグループは、形態や生殖的隔離及び生理的形質から7種(O.sativa,O.rufipogon,O.glaberrima,O.longistaminata,O.barthii,O.glumaepatula,O.meridionalis)に分類されている。しかし、これまでの形質による分類では、種名が頻繁に変更されたり、また異なる種とされた2種間で交配が可能であったり、問題点が残っている。O.sativaには、インディカ、ジャポニカ、ジャバニカという3つの生態種(ecospecies)に分けられているが、その判定基準は研究者によってまちまちであるため、各種の系統の近縁関係もはっきりせず、従って栽培稲の起源も異論がある。 1992年に梅田らはO.sativaのWaxy遺伝子のイントロンに125bpの挿入配列が存在していることを見出した。この挿入配列は、動物の染色体に存在するレトロポゾンであるSINE(short interspersed DNA elements)の特徴、即ちRNAポリメラーゼIIIのプロモーター配列(A-box,B-box)及び3’末端のT-richな配列を持つこと、標的配列の重複により生じた14bpのダイレクトリピートが両端に見られることから、植物(イネ)で初めて見つかったレトロポゾンとしてp-SINE1(plant-SINE#1)と名付けられた。1992年に望月らは、p-SINE1のメンバーの中には、O.sativaの系統と、祖先野生種であるO.rufipogonの一部の系統の特定の遺伝子座にのみ存在するものがあることを見出した。レトロポゾンはそれがゲノム内に一度挿入した場合、自分自身を切り出すという機能は持たない。従って、ある種においてp-SINE1が挿入している遺伝子座を同定し、他の種におけるそれぞれの遺伝子座についてのp-SINE1の挿入の有無を調べることによって、イネの分類が可能であると考えられた。 本研究はイネゲノム中に散在するいくつかのp-SINE1メンバーを分離し、塩基配列を決定することにより、それらの構造的特徴を明らかにすると共に、これらのp-SINE1メンバーを用いて、イネの系統の分類と近縁関係を明らかにし、イネの系統の分類上の混乱を解決することを目的として行ったものであり、結果は以下の様に要約できる。 1.p-SINE1メンバーの分離と同定(2,3) 本研究において、まずイネゲノム内に散在するp-SINE1のコピー数を測定し、O.sativaでは6500コピー存在することを明らかにした。この結果は、p-SINE1がO.sativaのゲノム上に平均70kbごとに存在することを示唆する。次に、異なる遺伝子座に存在する32個のp-SINE1メンバーを新たに分離し、塩基配列の解析をもとに、コンセンサス配列(122bp)を得た。各p-SINE1をコンセンサス配列と比較すると、多くの塩基置換変異が見られるばかりでなく、欠失・挿入・タンデム重複などの変異を生じたものがあることが分かった。 これらの各p-SINE1メンバーの隣接領域にハイブリダイズするプライマーを使用してPCRを行うと、唯一の例外を除き、単一のバンドのDNA断片が生じたことから、各メンバーはO.sativa染色体上のユニークな領域に存在すると考えられた。唯一の例外のp-SINE1の場合、PCR産物としてp-SINE1を含む断片と含まない断片が共に生じたが、この結果はイネゲノム中に2つの相同な遺伝子配列が存在しており、その片方にp-SINE1が挿入していると考えられた。さらに、得られたp-SINE1メンバーがAAゲノムを持つ様々な種の系統の各挿入部位に存在するか否か調べたところ、一部の種の全ての系統には存在しないメンバーや、特定の種の一部の系統には存在しないメンバーがあることが分かった。これらのp-SINE1メンバーの各挿入部位における存在の有無を調べることは、イネ系統の分類に利用可能であると考えられた。また、配列内に欠失・タンデム重複・挿入変異を持つp-SINE1において、変異が一部の種の全ての系統に存在するものや、特定の種の一部の系統に存在するものがあった。さらに、二つのp-SINE1メンバー(r32、r102)の3’端下流の隣接領域にはマイクロサテライトDNA、(CAT)nと(AG)n、が存在していたが、これらの配列の繰り返し数nがイネ系統により固有であることが分かった。p-SINE1メンバーにおける変異の存在の有無を調べること及び、マイクロサテライトDNAの反復数の違いを調べることによってもイネの系統の分類や栽培品種の識別に利用できる可能性が考えられた。 2.p-SINE1内の変異と新規転移性遺伝因子 Tnr3の発見(1,2) 得られたp-SINE1内部には塩基置換変異が数多くみられたが、これらのほとんどはG/CからA/Tへのトランジション変異によるものであった。また、数塩基対の欠失、タンデム重複、挿入等も起きていたが、その挿入・欠失変異の多くは同一塩基が連なる領域に起こっていた。さらに、大きなDNA領域の欠失、タンデム重複、挿入も見られた。特に挿入されている配列は後述するように転移性遺伝因子であることを見出した。このような変異は、p-SINE1のRNAポリメラーゼIIIのプロモーター内に生じていたことから、ほとんど全てのp-SINE1メンバーからのRNAポリメラーゼIIIによる転写は抑制されていると考えられる。 p-SINE1-r38メンバーに挿入していたTnr3(Transposable element in rice #3)と名付けた可動性遺伝因子1536bpは、3bpの標的配列を重複しており、5’-CACTA・・の配列で始まる13bpのIR(inverted repeat)を持っていた。また、IRの内側に隣接するサブターミナル領域には、15bpからなる異なる2つのモチーフが、35コピー順向き、及び、逆向きに存在した。このサブターミナル領域にはさらに異なる縦列型反復配列が5’側に3コピー、3’側に2コピー存在した。これらの構造的特徴は、トウモロコシのEn/Spmを始めとするCACTAファミリーと呼ばれる植物のトランスポゾンに見られるものと同様であった。しかし、Tnr3がこれらのトランスポゾンの特徴を持つものの、塩基配列全体の相同性は低く、実際にTnr3をプローブとして、トウモロコシ、大麦などの全ゲノムDNAを用いてサザンハイブリダイゼーションを行っても、シグナルは検出されなかったことから、イネに固有のトランスポゾンであると考えられた。また、Tnr3は全長1536bpという大きさから、転移酵素をコードする領域を持たない非自律性因子と推察された。実際、Tnr3の両端部分にハイブリダイズするプライマーを用いてPCRを行った結果、予想されるものよりも大きなPCR産物が得られ、この中にはTnr3の自律性因子が含まれると考えられる。 3.アジア栽培稲及び、近縁野生種の系統における分類と近縁関係 O.sativaと近縁野生種O.rufipogonを合わせ55系統(北陸農試の福田氏より分譲)とこれら以外の種の代表的な系統を用いて、分類に有用と思われるp-SINE1メンバー(r2、r6、r29、r30、r34)及びp-SINE1-r29とr30については内部の変異の有無、p-SINE1-r32については隣接領域に存在するマイクロサテライトDNAの(CAT)n反複数について、各エレメントの隣接領域にハイブリダイズするプライマーを用いてPCRによって調べた。それに加えて、O.sativaの系統間で挿入及び、欠失の有無がこれまでに確認されているマーカー、即ち、p-SINE1-r4に挿入していたトランスポゾンTnr2A、トランスポゾンTnr2Bの3’下流域に存在する欠失、特定の遺伝子座上のレトロトランスポゾンTos3、クロロプラストDNAのORF100内部の欠失、の有無についてPCRによって調べた。 表1に示したように、PCRによりp-SINE1の挿入を持つバンドを生じる系統(+)、それを生じない系統(-)、p-SINE1の挿入を持つバンドと持たないバンドの2つを生じる系統(+/-)が存在した。同一パターンを示す系統を1つのグループとした場合、O.sativaの系統は26グループ(A〜Z)に分けられ、他の種は、4グループ(a〜d)に分けられた。日本のジャポニカ型系統はすべて同一のパターンを示し、かなり均一な集団と考えられた。また、ジャバニカ型とされる系統は、採集場所が異なるにも関わらず同一のパターンを示した。インディカ型系統とO.rufipogonは、多くのグループに分かれ、遺伝的に多様な集団であることが示唆された。 表1を基にして、UPGMA法とNJ法によりデンドログラムの作製を試みた。その結果、O.sativa及びO.rufipogonの系統は、I〜VI群に分類された。この分類においては、ジャポニカ及びジャバニカも共にI群に属し、インディカはII群、III群、IV群に属し、O.rufipogonはIV、V群に多く属していた。この分類結果は、Glaszmannらのアイソザイム分析の結果と比較的一致するものであった。本研究で用いた方法は、1組の遺伝子座マーカーを使用しイネ系統、栽培品種をいわばバーコード化することによって分類し、近縁関係を知ることができる有用なものであると考えられる。 1)Motohashi R,Ohtsubo E and Ohtsubo H(1996)Identification of Tnr3, a Suppressor-Mutator/Enhancerlike transposable element from rice.Mol.Gen.Genet.1996 250:148-1522)Motohashi R,Mochizuki K,Ohtsubo E and Ohtsubo H(1996)Classification and relationships of rice strains by mobile genetic elements.International Rice Research Notes 1996 21:43)Motohashi R,Ohtsubo H and Ohtsubo E(1997)Structures and distribution of p-SINE1 members in rice genomes.Theor.Appl.Genet.in press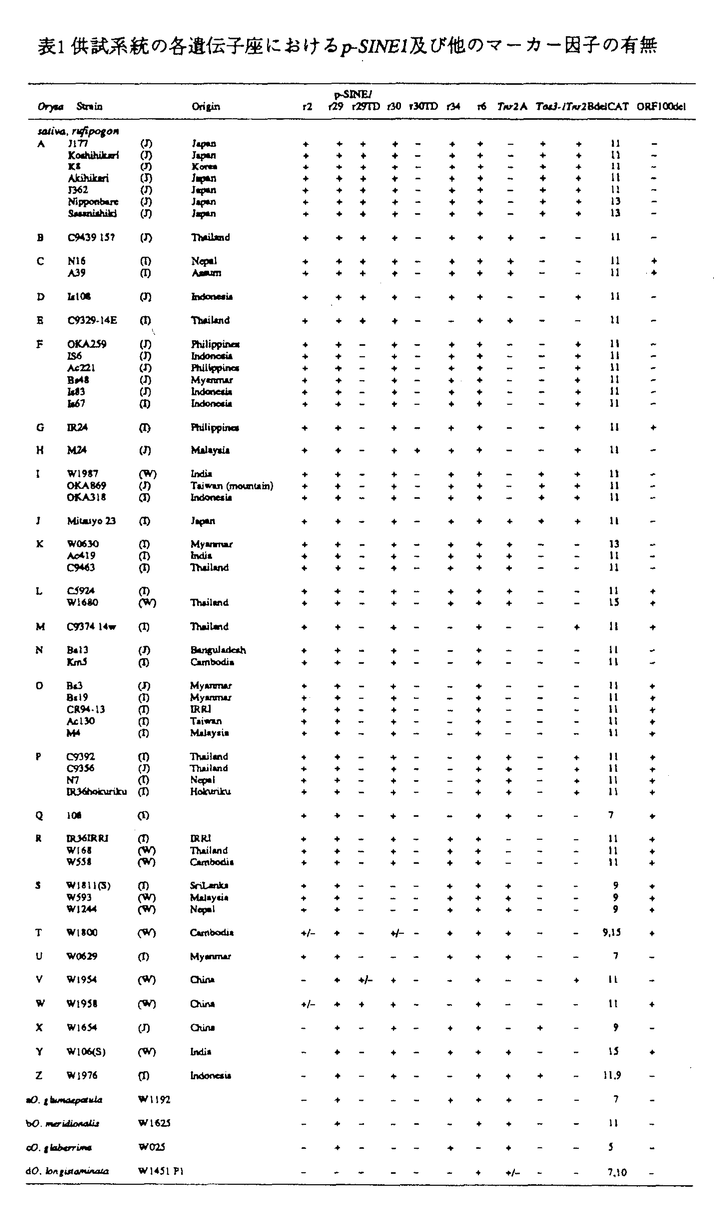 表1供試系統の各遺伝子座におけるp-SINE1及び他のマーカー因子の有無 表1供試系統の各遺伝子座におけるp-SINE1及び他のマーカー因子の有無 |