学位論文要旨
| No | 112739 | |
| 著者(漢字) | 川端,猛 | |
| 著者(英字) | ||
| 著者(カナ) | カワバタ,タケシ | |
| 標題(和) | 統計ポテンシャルを用いたタンパク質の構造予測 | |
| 標題(洋) | Protein structure prediction using statistical potential | |
| 報告番号 | 112739 | |
| 報告番号 | 甲12739 | |
| 学位授与日 | 1997.03.28 | |
| 学位種別 | 課程博士 | |
| 学位種類 | 博士(農学) | |
| 学位記番号 | 博農第1802号 | |
| 研究科 | 農学生命科学研究科 | |
| 専攻 | 応用生命工学専攻 | |
| 論文審査委員 | ||
| 内容要旨 | タンパク質の構造予測は構造生物学における依然未解決の難題である。その原因の一つはタンパク質の構成要素の複雑さであり、物理シミュレーションでアプローチする際には特に大きな問題となる。このような複雑な対象を扱う際のもう一つのアプローチとして、統計的アプローチ、すなわち、既知のデータの大域的な特徴が再現されるように単純なモデルを構築する方法がある。近年、データベースに登録されたタンパク質の結晶構造数は増加しており、タンパク質の構造予測においてもこのアプローチが有効となる可能性が高い。本研究では統計的アプローチの一種である「統計ポテンシャル(statistical potential)」と呼ばれる方法を試みた。このポテンシャルは構造の特徴をC、配列の特徴をAとしたとき、以下の式で記述される。 このポテンシャルはある配列aのときに構造cが平均より多く統計的に観察された場合負の値となる。全ポテンシャル値の最も小さい構造を予測構造とする。本研究では、このポテンシャルを2次構造予測と3次構造予測に適用し、その有効性・問題点を明らかにすることを目的とする。 タンパク質の2次構造予測は、立体構造予測の第一段階となる重要な技術であるが、十分高い予測精度を得る方法は未だ開発されていない。従来の2次構造予測法のほとんどは、X線結晶構造データベースから統計的に抽出した1残基ポテンシャル(値が1残基で決定されるポテンシャル)をもとに構成されている。1残基ポテンシャルは、アミノ酸ごとの2次構造傾向などを効率的に表現する、予測に不可欠な量であるが、予測精度を改善するためには、多残基ポテンシャル(値が複数残基によって決定されるポテンシャル)を取り入れる必要があると考えられる。しかしながら、多残基ポテンシャルを統計的に抽出するためには、結晶構造データベースの不足が大きな問題となる。例えば4残基ポテンシャルの場合でも、可能な4残基の全組合せ204=160,000は構造データベース内の残基数より遥かに多いため、統計有意な値を抽出することは困難である。 本研究ではアミノ酸を0,1の2種類に符号化することで、組合せの数を減らし有効に多残基ポテンシャルを抽出する方法を考えた。0,1に変換された局所配列を本研究では2元語(binary word)と呼ぶことにする。2元語の全パターン数は7残基対でもたかだか27=128に過ぎず、2元語自体を1つの事象と考えて多残基ポテンシャルを抽出することが可能である。本研究では1残基ポテンシャルを用いた古典的な方法であるGOR法に新たに2元語ポテンシャルを導入しその予測精度の改善を試みた。GOR法は以下のポテンシャルE(C=c:  このポテンシャルは、統計ポテンシャルの1種であり、式(1)を修正した形になっている。GOR法の1残基ポテンシャルの場合、式(2)の構造特徴C=cは中心残基の2次構造S0がsであることに対応し、配列の特徴A=aはm残基目のアミノ酸Amがaであることに対応する。GORの全ポテンシャルは以下のような1残基ポテンシャルの和で記述される。 本研究では新たに2元語ポテンシャルE(S0=s: このポテンシャルを用いた新しい予測法をBW-GOR法と呼ぶ。さらに、GOR法のポテンシャルに各2次構造ごとのオフセット値を加えたMGOR法、それに2元語ポテンシャルを組み合わせたBW-MGOR法を同様に作成した。 2元語符号を適用するには、まずアミノ酸の0,1への分類法を決定する必要がある。本研究ではシミュレーテツド・アニーリング法を用いて正答率を最大にする分類の探索を行った結果、次の分類法が最も正答率が高いことがわかった。 表1にこの分類法を用いた場合のシングル配列とマルチプルアライメント配列群の入力データに対する各方法の正答率を示す。2元語ポテンシャルを加えることで正答率は1.4〜2.8%改善した。マルチプルアライメント配列群を使用したBW-MGOR法の正答率がこの中では最高の68.2%であった。また2元語符号による正答率の改善は、多残基相互作用が2次構造形成に影響を及ぼすことを示唆する。さらに、探索で得られた式(5)の最適な符号化関数は明らかに非極性・疎水性を示していると考えられる。このことは多残基相互作用においては、疎水性相互作用が重要な役割を果たしていることを示唆する。 第II部では、統計ポテンシャルを3次構造予測に応用する。3次構造予測の場合、予測対象となる配座空間が極めて大きいため、いかにして簡潔で効率的なモデルを設計するかが重要な問題となる。以下に(a)構造表現、(b)ポテンシャル、(c)構造探索の3つに分けて本研究で開発した方法を説明する。 タンパク質の構造を表現する最小限のモデルとして、主鎖の二面角 式(1)の統計ポテンシャルを用いて作成する。いくつかのポテンシャルを試行した結果、以下の2つのポテンシャルを採用した。 ・距離ヒストグラムポテンシャル: Dij=dはi番目とj番目の ・局所構造ポテンシャル: Si=sはi番目の主鎖の二面角( これらのポテンシャルを、結晶構造データベースの構造群に配列をのせ、N構造がポテンシャル最小構造となっているかチェックするThreadingテストで評価した。2つを加算したポテンシャルEseq=EDab(d)+ELak(s)を用いた場合、74個中71個のタンパク質でN構造がポテンシャル最小構造となった。この結果はポテンシャルEseqが「タンパク質らしい」構造群からN構造を認識できる能力があることを示唆する。 Threadingテストの結果を受け、構造探索の目標を【与えられた配列のEseqが最も低い「タンパク質らしい」構造を探すこと】に設定する。本研究では「タンパク質らしい」構造の条件を(a)自己排除性 ここで、 Eseq、Eproの両方を用いて以下の手続きで予測構造を得る。 1.E=(1- 2.得られたポテンシャル最小構造群の中から、「タンパク質らしい」構造の条件を満たす構造を選択し、その中からEseqの最も低い構造を予測構造とする。 この予測法を80残基以下の5つのタンパク質に適用した。表2に得られた予測構造のポテンシャル   はN構造の、 はN構造の、 は予測構造の統計ポテンシャルを示す。RMSはN構造と予測構造の平均2乗誤差、QsecはN構造と予測構造で一致している2次構造の割合である。 は予測構造の統計ポテンシャルを示す。RMSはN構造と予測構造の平均2乗誤差、QsecはN構造と予測構造で一致している2次構造の割合である。 本研究では、統計ポテンシャルを2次構造予測と3次構造予測に適用した。2次構造予測においては新たに2元語ポテンシャルを加えることで予測精度を改善することができ、さらに、最適な符号化関数の探索から疎水性相互作用が2次構造形成に影響を及ぼすことが示唆された。3次構造予測においては、統計ポテンシャルEseqの低い「タンパク質らしい」構造を探索する方法を開発した。5つのタンパク質に適用した結果、得られた予測構造は、RMSは良い値ではなかったものの、2次構造は比較的良くN構造と一致し、タンパク質によっては全体のパッキングの様子もほぼ一致していた。今後、より精度の良い予測を行なうには、ポテンシャルや「タンパク質らしい」構造の条件を改善するとともに、実験的・進化的な情報を積極的に採り入れ、配座空間を狭めていく必要があると考えられる。 | |
| 審査要旨 | タンパク質の構造予測は構造生物学における依然未解決の難題である。タンパク質のような複雑な対象を扱う際のアプローチとして、統計的アプローチ、すなわち、既知のデータの大域的な特徴が再現されるように単純なモデルを構築する方法がある。本研究では統計的アプローチの一種である「統計ポテンシャル」と呼ばれる方法を採用し、このポテンシャルを2次構造予測と3次構造予測に適用している。 第1章では、統計ポテンシャルの理論的な考察を中心に、その有効性と問題点が的確にまとめられている。 第2章から第6章では、統計ポテシシャルの2次構造予測への適用について述べられている。従来の2次構造予測法の多くは、1残基ポテンシャル(値が1残基によって決定)をもとに構成されているが、予測精度を改善するためには多残基ポテンシャル(値が複数残基によって決定)を取り入れる必要がある。しかし多残基ポテンシャルの統計的な抽出は、構造データベースのサイズ不足による困難がある。第3章ではアミノ酸を0、1の2種類に符号化する「2元語符号」を導入している。2元語とは0、1に変換された局所配列のことであり、その全パターン数は少ないため、多残基ポテンシャルの統計的な抽出が容易であると考えられる。2元語ポテンシャルと古典的な予測法であるGOR法を組合わせたBW-GOR法、BW-MGOR法の2つを新たに導入した。第4章においては、まず分類を指定する符号化関数を決定するために、焼き鈍し法を用いて正答率を最大にする関数の探索を行っている。その関数を用いた2元語ポテンシャルを加えることで正答率は1.4〜2.8%改善した。マルチプルアライメント配列群を使用したBW-MGOR法の正答率がこの中では最高の68.2%であった。第5章では、物理化学的な符号化関数を用いた場合の正答率を比較し、非極性符号化関数の正答率が高いことを示した。第6章ではまとめとして、2元語符号による正答率の改善は多残基相互作用の2次構造形成への影響を示唆し、最適な符号化関数が非極性・疎水性を示していることは、疎水性相互作用が重要な役割を果たしていることを示唆していると結論づけている。 第7章から第11章では、統計ポテンシャルの3次構造予測への応用について述べられている。第7章には従来の3次構造予測法が簡潔にまとめられており、その一般性から、ポテンシャル最小化計算による予測が必要であることを主張している。そして、統計ポテンシャルでは陽に考慮されていない「タンパク質らしさ」を導入する必要性について述べている。第8章には予測法の詳細が述べられている。統計ポテンシャルEseqとして、距離ヒストグラムポテンシャル、局所構造ポテンシャルなどの数種の統計ポテンシャルが用意された。また構造探索の目標を【最もポテンシャルの低い「タンパク質らしい」構造を探すこと】に設定している。本研究では「タンパク質らしい」構造の条件を自己排除性、コンパクトさ、2次構造の形成の3つとし、この条件を満たすほど低くなるポテンシャルEproを設計している。そして、EseqとEproの和に対するポテンシャル最小化計算を繰り返すことにより、多数の局所最小構造を生成し、その中から「タンパク質らしい」構造を選択して、さらにその中からEseqの最も低い構造を予測構造としている。第9章では、まず最初に用意した統計ポテンシャルを、Threadingテストで評価している。距離ヒストグラムと局所構造ポテンシャルの和を用いた場合が最も成績が良く、ほとんどのタンパク質でN構造がポテンシャル最小となった。この予測法を80残基以下の5つのタンパク質に適用したところ、平均のRMSは11.3A、局所構造の一致率の平均は61.3%であり、局所構造はかなり一致しているものの、大域的構造には相違が見られた。第9章では、N構造との類似度とポテンシャルの相関の解析により、設定したポテンシャルの性質の吟味が行なわれている。第11章では、3次構造予測のまとめとして、本研究で開発した予測法では、「タンパク質らしさ」を考慮しない場合に比べ良好な予測結果を得たこと、予測構造は局所構造はよく一致したものの、大域的な構造には相違があったことが述べられている。大域的構造の相違の原因としてRMSとポテンシャルの相関の低さが指摘されている。 以上本論文は、統計ポテンシャルをタンパク質の2次構造予測と3次構造予測に適用した新しい方法を提案したものであり、学術上、応用上貢献するところが少なくない。よって審査委員一同は、本論文が博士(農学)の学位論文として価値あるものと認めた。 | |
| UTokyo Repositoryリンク | http://hdl.handle.net/2261/54586 |

 ;A=a)を基盤とする。
;A=a)を基盤とする。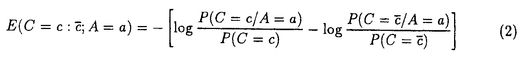

 ;W=w)を導入する。1残基ポテンシャルと同様に式(2)を用いて導出するが、配列の特徴A=aを2元語に変換された局所配列Wがwであることとする。この2元語ポテンシャルをGORのポテンシャルと加算した関数を新たに導入する。
;W=w)を導入する。1残基ポテンシャルと同様に式(2)を用いて導出するが、配列の特徴A=aを2元語に変換された局所配列Wがwであることとする。この2元語ポテンシャルをGORのポテンシャルと加算した関数を新たに導入する。

 ,
, で構造を記述し、
で構造を記述し、 原子と
原子と 原子のみを考慮するモデルを採用した。さらに配座空間を小さくするために(
原子のみを考慮するモデルを採用した。さらに配座空間を小さくするために(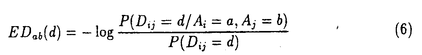
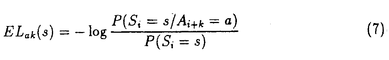
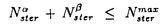 (b)コンパクトさ
(b)コンパクトさ
 (c)2次構造の形成
(c)2次構造の形成 の3つとした。そしてこの条件を満たすほど低くなるようなポテンシャルEproを以下のように設計した。
の3つとした。そしてこの条件を満たすほど低くなるようなポテンシャルEproを以下のように設計した。
 、
、 はそれぞれ衝突している
はそれぞれ衝突している はコンパクトな場合の慣性半径、Nhelix,Nsheetは
はコンパクトな場合の慣性半径、Nhelix,Nsheetは ヘリックス、
ヘリックス、 シートの残基数、Eregは局所構造の協同性を表すポテンシャルである。
シートの残基数、Eregは局所構造の協同性を表すポテンシャルである。 )Epro+
)Epro+