| 内容要旨 | | セントロメアは,真核生物の染色体分配時において中心的な役割を担う染色体領域である.一般にセントロメア領域のDNAは種によって異なる繰り返し配列から構成されており,通常のクロマチン構造とは異なる特殊な高次構造が形成されキネトコア形成の基盤となると考えられている.CENP-Bは哺乳類の染色体セントロメア領域において繰り返し配列で構成されるDNAの折り畳みに直接関与していると考えられているタンパク質である.したがって,セントロメア構造の構築原理を理解する上で,CENP-BのDNA認識機構に関する原子分解能での知見を得ることは重要なステップであるといえる.そこで,本研究では,主として多次元多核NMR技術を駆使して,ヒトCENP-Bおよびその分裂酵母でのホモログであるAbp1のDNA認識機構に関して構造生物学的解析をおこなった. CENP-BのDNA結合ドメイン(以下DBDと略称)はN端125残基に存在し,そのアミノ酸配列から二つのリピート構造RP1,RP2から構成されるのではないかと予測されていた.まず本研究では,RP1の解析をおこなった.RP1の大量発現系を構築し,得られたタンパク質を用いて熱変性曲線の解析をおこない実際にRP1がそれ自身安定な構造を形成するドメインであることを確認した.さらに1H,13C,15N多次元多核NMRを用いてCENP-B DBD RP1の立体構造を決定した.その結果,RP1は4本の ヘリックスをもち,ヘリックス-ターン-ヘリックス構造(以下HTH構造と略称)を形成することが明らかになった.アミノ酸配列の相同性はないが,helix1からhelix3まではホメオドメイン,Mybのリピート単位,テロメア結合タンパクRAP1 DBDのdomain1,2と類似している. ヘリックスをもち,ヘリックス-ターン-ヘリックス構造(以下HTH構造と略称)を形成することが明らかになった.アミノ酸配列の相同性はないが,helix1からhelix3まではホメオドメイン,Mybのリピート単位,テロメア結合タンパクRAP1 DBDのdomain1,2と類似している.  図1 CENP-B DBD RP1の立体構造のリボン図 図1 CENP-B DBD RP1の立体構造のリボン図 さらにCENP-B DBD RP1とCENP-BボックスをふくむDNAとの相互作用をNMR化学シフト変化を用いて解析した.この際にもちいたDNAはCENP-B DBD全体(RP1-RP2)と結合できる最小DNAフラグメント(CB21)である.CENP-B DBD RP1上でのDNA結合部位を調べるために,15N標識されたCENP-B DBD RP1にCB21を混合し,2D1H-15N HSQCスペクトルの変化を調べた.その結果,RP1はN端部分の塩基性領域,helix2のN端側,helix3でDNAと結合することがわかった.またDNA上でRP1が結合する配列を調べるために,RP1の存在下,非存在下でのイミノプロトンの化学シフト変化を調べた(図2).変化の大きい領域は,CENP-B DBDとの結合に必要な3つの領域のうちのひとつ,site 1とよく重なる.したがってCENP-B DBD RP1はsite 1に結合すると考えられる.また,のこりのsite2,site3に関してはそれぞれリンカーおよびRP2のHTH構造が結合すると考えるのが妥当である.これらの結果をもとにCENP-B DBD RP1-RP2全体とCENP-BボックスDNAの複合体モデルを考察した(図3).このモデルは,RP1のhelix3とhelix4がなす角度は短いリンカーで連結されたRP1およびRP2のHTH構造がともにDNAに接触するのに重要であることを示唆している. 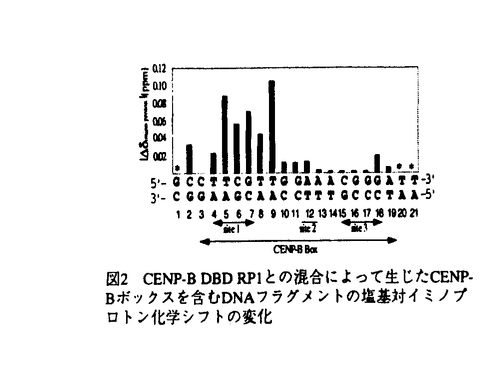 図2 CENP-B DBD RP1との混合によって生じたCENP-Bボックスを含むDNAフラグメントの塩基対イミノプロトン化学シフトの変化 図2 CENP-B DBD RP1との混合によって生じたCENP-Bボックスを含むDNAフラグメントの塩基対イミノプロトン化学シフトの変化 図3 CENP-B DBD(RP1-RP2)とCENP-BボックスDNAとの複合体モデル 図3 CENP-B DBD(RP1-RP2)とCENP-BボックスDNAとの複合体モデル ただ,これはあくまでもモデルであり,詳細に関して実験的に検証する必要性がある.しかしながら,CENP-B DBDとDNAの複合体は安定性,溶解度の点で問題があり,NMRで解析できる条件がみつからなかった.そこで,分裂酵母でのCENP-BのホモログであるAbp1に注目し,そのDBDとDNAの複合体の解析を試みた.本研究以前はAbp1のDBDは同定されていなかったが,Abp1はCENP-Bとアミノ酸類似性が50%ほどあり,CENP-BのDBDと相同性のある領域がAbp1のDBDであると考えられた.そこでそのような領域にあたるAbp1のN末端141残基の大量発現型を構築し,Abp1の認識配列を含むDNA(AM21)との結合を調べた.実際,このAbp1のフラグメントがAbp1認識配列に対して結合能を有することが確認できた(図4).以後,この領域をAbp1 DBDとよぶ.AM21DNAフラグメントに対するAbp1 DBDのモル濃度比が0,0.7,1.0の混合系に関するDNAの塩基対イミノプロトンの1D1Hスペクトルの比較(図5)から,Abp1 DBD・DNA複合体の解離はNMR化学シフトタイムスケールに対して遅い交換過程であること,Abp1は認識配列に対して1:1で結合することがわかる.分子量17kDaのAbp1 DBDと13kDaのAM21 DNAフラグメントから構成される総分子量30kDaのこの複合体に関してNMR解析をおこなった.  図4 Abp1 DBDと認識配列を含むDNAとの結合 図4 Abp1 DBDと認識配列を含むDNAとの結合 図5 異なる[Abp1 DBD]/[AM21 DNA]モル比での1D1HスペクトルのDNAイミノプロトン領域 図5 異なる[Abp1 DBD]/[AM21 DNA]モル比での1D1HスペクトルのDNAイミノプロトン領域 図6はAbp1 DBD・DNA複合体の2D1H-15N HSQCスペクトルである. ヘリックスが多いタンパク質としては,ピークが良好に分離している.Abp1 DBD・DNA複合体のNMR解析に際しては,大きな分子量に起因する短いNMR緩和時間によるスペクトル感度の低下を防ぐため,Abp1 DBDをランダムに-60%重水素化し,2Hデカップリングを組み込んだ各種1H,13C,15N多次元多核NMR測定をおこなった. ヘリックスが多いタンパク質としては,ピークが良好に分離している.Abp1 DBD・DNA複合体のNMR解析に際しては,大きな分子量に起因する短いNMR緩和時間によるスペクトル感度の低下を防ぐため,Abp1 DBDをランダムに-60%重水素化し,2Hデカップリングを組み込んだ各種1H,13C,15N多次元多核NMR測定をおこなった.  図6 Abp1 DBD・DNA複合体の2D1H-15N HSQCスペクトル.DNAは標識されていないので,Abp1 DBD側のシグナルだけが観測される.またAbp1 DBDは〜60%重水素化されている. 図6 Abp1 DBD・DNA複合体の2D1H-15N HSQCスペクトル.DNAは標識されていないので,Abp1 DBD側のシグナルだけが観測される.またAbp1 DBDは〜60%重水素化されている. これらのスペクトルを解析することにより,複合体中のAbp1 DBD主鎖13C ,13CO,15N,1H ,13CO,15N,1H ,1HNの化学シフトの帰属を完了し,帰属可能なほとんど全ての側鎖1H,13C,15N化学シフトの帰属を完了した.また,short,medium rangeのNOEから複合体中のAbp1 DBD RP1の二次構造を決定した(図7).Abp1 DBDは全部で7本の ,1HNの化学シフトの帰属を完了し,帰属可能なほとんど全ての側鎖1H,13C,15N化学シフトの帰属を完了した.また,short,medium rangeのNOEから複合体中のAbp1 DBD RP1の二次構造を決定した(図7).Abp1 DBDは全部で7本の ヘリックスをもつ.CENP-B DBD RP1と相同性のあるMet1からGlu66までにhelix1からhelix4までが含まれ,そのパターンからこの領域はCENP-B DBD RP1と類似したHTH構造をもつドメインだと考えられる.また残りの,Lys67からHis141までにhelix5からhelix7までの3本の ヘリックスをもつ.CENP-B DBD RP1と相同性のあるMet1からGlu66までにhelix1からhelix4までが含まれ,そのパターンからこの領域はCENP-B DBD RP1と類似したHTH構造をもつドメインだと考えられる.また残りの,Lys67からHis141までにhelix5からhelix7までの3本の ヘリックスが存在するので,この領域もHTH構造をもつドメインである可能性が高く,Abp1は二つのHTH構造ドメインで形成されると考えられる. ヘリックスが存在するので,この領域もHTH構造をもつドメインである可能性が高く,Abp1は二つのHTH構造ドメインで形成されると考えられる.  図7 Abp1 DBD・DNA複合体中におけるAbp1 DBDで観測されたshort,medium range NOEおよび決定した二次構造.また複合体サンプルのNaCl濃度を0mMから200mMに変化させた時にブロードニングした残基を▲で示した. 図7 Abp1 DBD・DNA複合体中におけるAbp1 DBDで観測されたshort,medium range NOEおよび決定した二次構造.また複合体サンプルのNaCl濃度を0mMから200mMに変化させた時にブロードニングした残基を▲で示した. Abp1 DBD・DNA複合体中でのAbp1 DBDのDNA結合部位を調べるため,イオン強度上昇により複合体を解離させ,その過程での複合体の2D1H-15N HSQCスペクトルの変化を調べた.NaCl濃度を0mMから200mMに変化させると,一部のピークがブロードニングを起こした.これは複合体の結合が弱まり,解離がNMR化学シフトタイムスケールに対して,いわゆる"intermediate exchange"に近くなったため,複合体中および単体中での化学シフトの差が大きいシグナルがブロードニングを起こしたと考えられる.複合体中および単体中での化学シフトの差が大きい残基は,DNAの相互作用部位である可能性が高い.実際,同位体フィルター測定により分子間NOEのクロスピークが観測された残基の多くが,この実験でブロードニングを生じた残基であった.したがって,この実験でシグナルがブロードニングした残基を調べることにより,複合体中でDNAと相互作用している残基をマッピングすることができる.図7で▲で示された残基はブロードニングがみられた残基である.CENP-B DBD RP1と相同性のある領域は,これらの残基の分布がCENP-B DBD RP1のDNAへの結合部位の分布に類似している.したがってAbp1とCENP-Bは少なくともRP1に相当する部分に関してはDNAへの結合様式が類似している可能性が高い.また,ふたつのドメインのリンカー部分やhelix7にも多くブロードニングを起こす残基がある.したがって,Abp1 DBDに存在すると考えられる二つのドメインおよびリンカーが共にDNA結合に関わっていると考えられる. |
| 審査要旨 | | セントロメアは真核生物の染色体分配時に中心的な役割を果たす染色体領域である.その領域のDNA鎖は一般的に生物種によって異なる反復配列から構成されれる.これらの反復配列がセントロメアに局在するタンパク質によって高度に折り畳まれ,それが動原体(キネトコア)形成の基盤となっていると考えられている. 本論文では,このようなセントロメア領域の折り畳みに重要な機能を果たしていると考えられているCENP-Bファミリーに注目し,ヒトCENP-Bおよびその分裂酵母におけるホモログであるAbp1のDNA結合ドメインに関してNMRを駆使した構造生物学的解析をおこなっている. 第2章では,ヒトCENP-BのDNA結合ドメインRP1の構造解析に関して述べられている.まず,NMRを用いて明らかにされたRP1の立体構造に関して記述されている.これはセントロメア結合タンパク質として,初めて立体構造が明らかにされた例である.RP1は4本の ヘリックスを有し,helix2からhelix3がヘリックス-ターン-ヘリックス構造を形成する.RP1のhelix1からhelix3に関しては特にテロメア結合タンパク質RAP1のDNA結合ドメインと類似の構造であることが示されている.また,DNA結合部位の局在パターンがホメオドメイン,Myb,RAP1のDNA接触部位の局在パターンと類似していることから,CENP-BのRP1はこれらのタンパク質DBDと同様にhelix3を認識ヘリックスとしてDNAと相互作用すると考察される.さらにDNA塩基対イミノプロトンの化学シフト変化の解析から,RP1がCENP-Bの結合配列CENP-Bボックスのsite1を認識することを示し,その結果をふまえてCENP-BのDNA結合ドメイン全体とDNA(CENP-Bボックス)の複合体モデルを提示している. ヘリックスを有し,helix2からhelix3がヘリックス-ターン-ヘリックス構造を形成する.RP1のhelix1からhelix3に関しては特にテロメア結合タンパク質RAP1のDNA結合ドメインと類似の構造であることが示されている.また,DNA結合部位の局在パターンがホメオドメイン,Myb,RAP1のDNA接触部位の局在パターンと類似していることから,CENP-BのRP1はこれらのタンパク質DBDと同様にhelix3を認識ヘリックスとしてDNAと相互作用すると考察される.さらにDNA塩基対イミノプロトンの化学シフト変化の解析から,RP1がCENP-Bの結合配列CENP-Bボックスのsite1を認識することを示し,その結果をふまえてCENP-BのDNA結合ドメイン全体とDNA(CENP-Bボックス)の複合体モデルを提示している. 第3章では,CENP-BファミリーのDNA結合様式を実験的に明らかにするために分裂酵母におけるCENP-BホモログであるAbp1のDNA結合ドメインとその認識DNAとの複合体を研究対象としておこなった解析に関して記述されている.まず,CENP-B DNA結合ドメインと相同性をもつAbp1のN端141残基が実際にDNA結合活性をもち,認識DNA1分子に対して1分子のAbp1が結合することをゲルシフト法およびNMRを用いた結合実験により示している.次に,分子量3万のこの複合体に関して,様々な多次元NMR技術を駆使しておこなった構造解析に関して論じている.複合体中におけるAbp1の主鎖および側鎖のNMRシグナルを帰属し,二次構造を決定している.Abp1 DNA結合ドメインAbp1のDBDは7本の ヘリックスを形成し,うち最初の4本がCENP-B DNA結合ドメインRP1と類似のパターンをしめす.このことからAbp1 DNA結合ドメインはCENP-Bで考察されたようにふたつのドメイン構造から形成され,うちN端側のドメインに関してはCENP-B DBD RP1と類似した立体構造を形成すると結論できる.また,同位体フィルター測定による分子間NOEの測定,イオン強度変化による複合体スぺクトルの変化の解析などから,DNAとの相互作用残基を解析し,ふたつのドメイン両方がDNAと相互作用しているという結論が導かれる. ヘリックスを形成し,うち最初の4本がCENP-B DNA結合ドメインRP1と類似のパターンをしめす.このことからAbp1 DNA結合ドメインはCENP-Bで考察されたようにふたつのドメイン構造から形成され,うちN端側のドメインに関してはCENP-B DBD RP1と類似した立体構造を形成すると結論できる.また,同位体フィルター測定による分子間NOEの測定,イオン強度変化による複合体スぺクトルの変化の解析などから,DNAとの相互作用残基を解析し,ふたつのドメイン両方がDNAと相互作用しているという結論が導かれる. 以上の研究は,すべて本論文提出者が主体となって研究計画,実験,検証をおなったものであり,審査委員会は本論文提出者が博士(理学)の学位を受ける資格あるものと判定した.なお,本論文第2章は東京大学の横山茂之教授,名古屋大学の岡崎恒子教授(現藤田保健衛生大学),舛本寛博士,北川克己博士,理化学研究所の木川隆則博士との共同研究であるが,論文提出者が主体となって分析および検証をおこなったものなので,論文提出者の寄与が充分であると判断した. |