学位論文要旨
| No | 114424 | |
| 著者(漢字) | 堀之内,正枝 | |
| 著者(英字) | ||
| 著者(カナ) | ホリノウチ,マサエ | |
| 標題(和) | 微生物によるジメチルスルフィドの酸化 | |
| 標題(洋) | ||
| 報告番号 | 114424 | |
| 報告番号 | 甲14424 | |
| 学位授与日 | 1999.03.29 | |
| 学位種別 | 課程博士 | |
| 学位種類 | 博士(農学) | |
| 学位記番号 | 博農第2032号 | |
| 研究科 | 農学生命科学研究科 | |
| 専攻 | 応用生命工学専攻 | |
| 論文審査委員 | ||
| 内容要旨 | ジメチルスルフィド(dimethyl sulfide;DMS)は海洋から多量に発生していることが知られており,二酸化硫黄についで大気中に多く存在する硫黄化合物として,地球規模での硫黄循環において重要な役割を果たしていると考えられている化合物である。DMSは大気中に放出される前にその90%あまりが海洋中で微生物等によって変換されるとする報告もあり,また様々な微生物がDMS変換能を有することが報告されていることから,DMSの変換において微生物が果たす役割は重要であると考えられている。しかし,DMS変換に関与する微生物についての知見は,一部のC1化合物資化性菌を除けば不十分といわざるを得ず,特にDMS変換を行う酵素遺伝子に関する報告はまったくなされていなかった。 DMS変換に関与する微生物の報告は海洋由来のものが多いが,当研究室でこれまでに行われた研究からは,DMSを硫黄源として生育可能な菌株が土壌中にも多数存在し,これら菌株の多くはDMSを最初にジメチルスルホキシド(dimethyl sulfoxide;DMSO)に変換することが示唆されていた。この最初の反応はDMSを硫黄源として生育可能な菌株のDMS代謝において最も重要と考えられることから,DMS酸化酵素遺伝子の取得が試みられてきたが,これまでDMS酸化活性の効率的な検出方法が確立しておらず,当該酵素遺伝子の取得には至っていなかった。 そこで,本研究ではDMS酸化酵素遺伝子のスクリーニングにインドールの共酸化の結果生じるインジゴの呈色を利用することを考え,DMS酸化能とインドール酸化能を併せ持つ菌株を単離し,その菌株からDMS酸化酵素遺伝子の取得を試み,微生物によるDMSの変換に関する基礎的知見を得ることを目的とした。 これまで当研究室で単離されたDMS資化菌にインドールの酸化能は認められなかったことから,はじめにインドール酸化能,DMS資化能,およびDMSのDMSOへの酸化能を有する菌株の取得を行った。その結果取得されたAcinetobacter sp.20B株は,DMSを硫黄源として培養すると培養上清に多量のDMSOが蓄積する事が明らかとなった。この20B株のtotal DNAをXbaI処理して得られたゲノムライブラリーから,インジゴの呈色を示す大腸菌形質転換体が取得された。この形質転換体を用いてDMSを基質に休止菌体反応を行った結果,多量のDMSOが生成することが確認された。この形質転換体の保持するプラスミドを抽出してDMS酸化活性に必須な領域を含むデリーションプラスミドpAU96を作製,挿入断片の塩基配列を決定したところ,この領域には6個のORFが同じ向きにコードされていることが明らかとなった。これらORFの推定アミノ酸配列は,multicomponent phenol hydroxylaseであるMopKLMNOP,DmpKLMNOP等と高い相同性を示したことから,これらをDsoABCDEFと命名した(Fig.1)。またDsoDとDsoFでは活性中心と考えられるモチーフが保存されていた。  DsoABCDEFの各々のタンパク質の機能が対応するDmpKLMNOPの各々の機能と同様であることを確認するため,各々のタンパク質一つずつが欠失したプラスミドを構築し,それらを保持する大腸菌のDMS酸化活性を調べた。DsoABCDEF全てが低コピー数のpSTV28(Cmr)にコードされているpAS96の場合はDMSOの収率は約30%であり,DsoBCDEFが各々欠失しているpASB   〜F 〜F (pSTV28/29由来,DsoA〜Fがそれぞれ欠失),カラム3:pASA (pSTV28/29由来,DsoA〜Fがそれぞれ欠失),カラム3:pASA 〜F 〜F +pAUA〜F(pUC118/119由来,DsoA〜Fそれぞれをコード),カラム4:pASA +pAUA〜F(pUC118/119由来,DsoA〜Fそれぞれをコード),カラム4:pASA 〜F 〜F +pVI281 +pVI281 〜221 〜221 (pMMB (pMMB  EH/HE由来,DmpK〜Pそれぞれをコード) EH/HE由来,DmpK〜Pそれぞれをコード) 今回DMS酸化活性を有する酸化酵素として単離されたDsoABCDEFは従来phenol hydroxylaseとして単離されていた多くの酸化酵素と極めて類似しており,phenol hydroxylaseを含め,他の既知の芳香族化合物酸化酵素もDMS酸化活性を有する可能性が考えられた。そこで,DsoABCDEFと類似の遺伝子構造を有するDmpKLMNOP,AphKLMNOP,TbmABCDEF,single component phenol hydroxylase(TfdB),multicomponent dioxygenaseであるcumene dioxygenase(CumA1A2A3A4),toluene-xylene dioxygenase(XylMA),naphthalene-phenanthrene dioxygenase(PahAaAbAcAd),carbazole dioxygenase(CarAaAcAd),およびdibenzothiophene dioxygenase(SoxC)の各種酸化酵素の,DMSからDMSOへの酸化活性を調べた。この際,必要があればベクターの組換えを行って,酸化酵素は全てpUC18/19,またはpUC118/119から発現されるようにした。DsoABCDEFと類似の遺伝子構造を有するDmpKLMNOP,AphKLMNOP,TbmABCDEFはDMSの酸化活性を有することが明らかとなったが,AphKLMNOP,TbmABCDEFの活性はDsoABCDEF,DmpKLMNOPと比較し非常に低かった(Fig.4)。Single component phenol hydroxylaseのTfdBにはDMS酸化活性はほとんど見いだされなかった。その一方で,multicomponent dioxygenaseであるCumA1A2A3A4,XylMA,PahAaAbAcAdにもDMS酸化活性が認められた。Dibenzothiopheneの硫黄原子に対する酸化活性を有する事からDMSの酸化活性も期待されたCarAaAcAd,SoxCにはDMSの酸化能は認められなかった。これらの結果より,遺伝子構造の類似性とは無関係に,様々な酸化酵素がDMS酸化活性を有している可能性が示された。 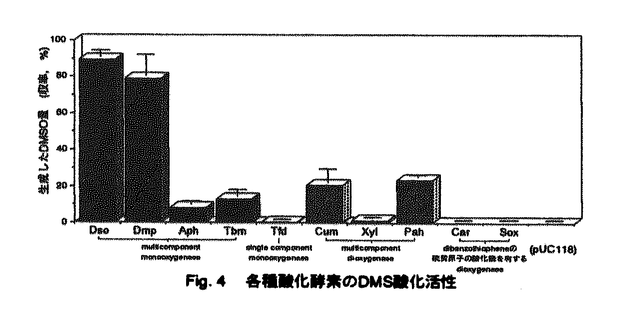 本研究では土壌からDMS資化菌Acinetobacter sp.20B株を単離すると共に,20B株からDMS酸化活性を有する酵素遺伝子dsoABCDEFの単離と解析を行い,DsoABCDEFはDmpKLMNOP等,multicomponent phenol hydroxylaseと高い相同性を示すことを明らかにした。DmpKLMNOPとの相補実験からは,DsoABCDEFの各タンパク質は対応するDmpKLMNOPの各タンパク質と同様の機能を有することが示唆された。また,本研究によって様々な酸化酵素がDMS酸化活性を有することが示された。今回DMS酸化活性を有することが明らかとなった酸化酵素には全てインドールの酸化活性が認められたこと,また,本実験でDMS資化菌として単離された多数の菌株のうちインドール酸化能を有する菌株はごく少数であったことから,DsoABCDEFのように芳香族化合物にも酸化活性を有する酸化酵素とは異なるタイプのDMS酸化酵素が多く存在するものと考えられる。DMS変換における微生物の果たす役割を解明するためには,今後はこれら未同定のDMS酸化酵素遺伝子を解析していくことが必要と考えられる。 | |
| 審査要旨 | 本研究は、地球規模での硫黄循環において鍵となる化合物であるジメチルスルフィド(DMS)の微生物変換に関する知見を得ることで地球規模での硫黄循環の理解に寄与することを目的とし、新規DMS資化菌Acinetobacter sp.20B株からDMSのDMSOへの酸化活性を有する酵素遺伝子dsoABCDEFを取得し、DmpKLMNOPの各タンパク質を用いた相補実験を行って各タンパク質の機能を推定するとともに、微生物由来の様々な酸化酵素のDMS酸化活性の検討を行ったものであり、全4章からなる。 第1章の序論に引き続き、第2章では、これまで適切な検出方法が見いだせなかったためにクローン化に至っていなかったDMS酸化酵素遺伝子の取得方法として、酸化酵素のクローニング法のひとつとして知られるインジゴ発色を利用する方法を用いるため、DMSを硫黄源として生育し、インドール酸化活性とDMSのDMSOへの酸化活性をあわせ持つDMS資化菌Acinetobacter sp.20B株を土壌より単離した。この際、DMSをDMSOを経由して硫黄源として資化できる菌株は土壌より非常に多数、高頻度で単離された。このことは、環境中で微生物によるDMSのDMSOへの酸化は普遍的に起こっているとの推測を支持するものと考えられた。また、20B株はジエチルスルフィド、ジメチルジスルフィド等も硫黄源として生育できることが見いだされた。 第3章においては、第2章で単離した20B株からDMSのDMSOへの酸化活性を有する酵素遺伝子dsoABCDEF遺伝子を取得して塩基配列の決定、遺伝子解析を行った。DsoABCDEFは、フェノールヒドロキシラーゼであるAcinetobacter calcoaceticus NCIB8250株のMopKLMNOPやPseudomonas sp.CF600株のDmpKLMNOPと高い相同性を示すことが明らかとなったので、類似の酸化酵素の中で最も解析の進んでいるDmpKLMNOPの各タンパク質を用いた相補実験を行ってDsoABCDEFの各タンパク質の機能を推定した。相補実験の前に各タンパク質を欠失させ、DsoABCDEFの大腸菌をホストとした場合のDMS酸化活性にはDsoAは不要であることを示した。相補実験の結果からは、DsoBDEがオキシゲナーゼコンポーネント、DsoFがレダクターゼ、DsoAがアポ酵素に鉄イオンを挿入するタンパク質、DsoCが活性化タンパク質と推測された。また、今回の相補実験の際に、オキシゲナーゼコンポーネントを構成するサブユニットであるDsoBDEの結合のための認識は他のタンパク質に比べ厳密であることが示され、一方でDsoBDE間の結合の厳密性には差がある可能性も示唆された。その他に、大腸菌内でDsoABCDEFを発現させた場合には、DsoFの発現量が活性の律速となっている可能性が示された。 第4章においては、DsoABCDBFがフェノールヒドロキシラーゼと高い相同性を示したことから、各種芳香族化合物酸化酵素のDMS酸化活性の検討を行い、ジオキシゲナーゼを含めた構造の全く異なる酸化酵素にもDMS酸化活性が存在することを示した。また、用いた酸化酵素の多くにDMS酸化活性が見いだされたことは、自然界では様々な酸化酵素がDMS酸化を担っているとの予測を支持するものと考えられた。 以上、本論文は、DMS酸化活性を有する酵素遺伝子dsoABCDBFを初めて単離し、DsoABCDEF各タンパク質の機能を推測すると共に、遺伝子構造の異なる各種芳香族化合物酸化酵素がDMS酸化活性を有することを見いだす等、地球規模での硫黄循環の鍵となるDMSのDMSOへの微生物変換に関する新知見を与えたものとして学術上、応用上貢献するところが少なくない。よって、審査委員一同は、本論文が博士(農学)の学位論文として価値あるものと判断した。 | |
| UTokyo Repositoryリンク | http://hdl.handle.net/2261/54708 |