本研究はヒトIL-3およびGM-CSFの特異的生理活性は各レセプター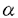 鎖(hIL-3R 鎖(hIL-3R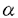 ,hGMR ,hGMR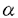 )の発現パターンに反映するとの考えから、 )の発現パターンに反映するとの考えから、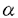 鎖遺伝子の転写レベルでの発現調節機構に着目し、それに関与する転写因子の同定を目的としたものであり、以下の結果を得ている。 鎖遺伝子の転写レベルでの発現調節機構に着目し、それに関与する転写因子の同定を目的としたものであり、以下の結果を得ている。 1.両遺伝子の細胞特異性をRT-PCRを用いてmRNAレベルで解析した結果、両者ともTF-1とEoL3に対して発現し、JurkatとHeLaに発現していないことや、また、hGMR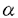 が発現し、hIL-3R が発現し、hIL-3R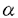 は発現していないことがU937で認められた。以上の結果はこれらの は発現していないことがU937で認められた。以上の結果はこれらの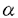 鎖遺伝子が細胞種特異的な発現調節を受けること、hIL-3R 鎖遺伝子が細胞種特異的な発現調節を受けること、hIL-3R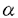 とhGMR とhGMR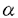 では異なる機構によって発現が調節されていることが示された。 では異なる機構によって発現が調節されていることが示された。 2.単離したhGMR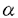 遺伝子プロモーター領域を約3kbまで遺伝子配列を決定し、そのうち-2449から-10までの範囲で様々なdeletion mutantを作成し、TF-1においてLuciferse assayによる転写活性化領域を同定したところ、-53から-11のPU.1結合配列を含む領域が転写活性の促進に、その上流約1kbにおよぶTG-rich配列が抑制に関係することが示唆された。また、-53から-11の領域はTF-1とU937に対し細胞特異的に転写活性を促進することも示された。 遺伝子プロモーター領域を約3kbまで遺伝子配列を決定し、そのうち-2449から-10までの範囲で様々なdeletion mutantを作成し、TF-1においてLuciferse assayによる転写活性化領域を同定したところ、-53から-11のPU.1結合配列を含む領域が転写活性の促進に、その上流約1kbにおよぶTG-rich配列が抑制に関係することが示唆された。また、-53から-11の領域はTF-1とU937に対し細胞特異的に転写活性を促進することも示された。 3.Western blottingによってTF-1にPU.1が存在することを確認した後、PU.1結合配列が含まれる領域のDNA断片を用い、Gel shift assayを行ったところ、この領域に特異的に結合する核蛋白が結合することが示された。またSupershift assayによって、この核蛋白がPU.1であることを示したが、このPU.1は並行して行われたU937でのそれと異なるサイズであることも示された。 4.IL-3R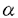 遺伝子のプロモーター領域を同定するため、Primer extension法および5’-RACEによって転写開始点を決定した。結果、翻訳開始点から256bpから269bp上流にかけて転写開始点が複数存在することが示され、両実験において一致した点として翻訳開始点から267bp上流のTを転写開始点の+1と定めた。 遺伝子のプロモーター領域を同定するため、Primer extension法および5’-RACEによって転写開始点を決定した。結果、翻訳開始点から256bpから269bp上流にかけて転写開始点が複数存在することが示され、両実験において一致した点として翻訳開始点から267bp上流のTを転写開始点の+1と定めた。 5.hIL-3R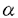 遺伝子プロモーター領域を約1.4kbまで遺伝子配列を決定し、その領域を-49までの範囲で様々なdeletion mutantを作成し、hGMR 遺伝子プロモーター領域を約1.4kbまで遺伝子配列を決定し、その領域を-49までの範囲で様々なdeletion mutantを作成し、hGMR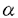 と同様、TF-1においてLuciferse assayによる転写活性化領域を同定したところ、-363から-331および-124から-76の領域が転写活性の促進に関係することが示唆された。またその他に、-330から-273の領域がプロモーター活性を抑制することや、hGMR と同様、TF-1においてLuciferse assayによる転写活性化領域を同定したところ、-363から-331および-124から-76の領域が転写活性の促進に関係することが示唆された。またその他に、-330から-273の領域がプロモーター活性を抑制することや、hGMR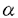 とは対照的にhIL-3R とは対照的にhIL-3R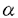 プロモーター領域に含まれる全てのPU.1結合配列はプロモーター活性の促進に関与しないことなども示された。しかしながら、この領域における転写活性に細胞特異性は認められなかった。 プロモーター領域に含まれる全てのPU.1結合配列はプロモーター活性の促進に関与しないことなども示された。しかしながら、この領域における転写活性に細胞特異性は認められなかった。 6.-124から-76の領域に結合する転写因子の存在を明らかにするため、Gel shift assayおよびCompetition analysisを行ったところ、-106から-92の領域内に2種類の核蛋白が特異的に結合することが示された。さらにSupershift assayにおいて、その中の1種類がSp1であることが示された。また-363から-331の領域においても同様の実験を行ったところ、複数の核蛋白群がこの領域への結合に関与することが示された。また、この領域の塩基配列は現在までデーターベースなどで該当する転写因子が存在しないため、新規の転写因子が関与していることが示唆された。 以上、本研究はhIL-3R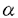 およびhGMR およびhGMR のプロモーター領域の解析により、両者の遺伝子発現がそれぞれ独立した転写因子によって調節を受けていることを明らかにした。本研究は各 のプロモーター領域の解析により、両者の遺伝子発現がそれぞれ独立した転写因子によって調節を受けていることを明らかにした。本研究は各 鎖の発現パターンがIL-3およびGM-CSFの特異的生理活性に反映すると考えられる。またサイトカインに対する細胞の応答性の解明に重要な貢献をなすと考えられ、学位の授与に値するものと考えられる。 鎖の発現パターンがIL-3およびGM-CSFの特異的生理活性に反映すると考えられる。またサイトカインに対する細胞の応答性の解明に重要な貢献をなすと考えられ、学位の授与に値するものと考えられる。 |