ラン藻は自律的な運動能を獲得した最初の生物のうちの一つであり、そのメカニズムには運動能そのものの本質が現在に至るまで残されていると考えらているが、運動のメカニズムは未だに不明である。Synechocystis sp.PCC 6803は単細胞性のラン藻で光合成のモデル生物として広く研究がなされ、1996年にその全塩基配列が決定された。このPCC6803のパスツールカルチャーコレクションの元株は、運動性をもつ。申請者、岡本忍君はこの元株を用いて、ラン藻の運動メカニズムに関係する遺伝子の探索を行った。 1)配列決定株にのみ存在する挿入配列(IS) Synechocystis sp.PCC6803の運動能を有するPCC6803の元株にトランスポゾン、ランダムカセット挿入などにより突然変異誘発し、運動しない株のスクリーングすること試みた。その解析過程において興味深い発見をした。ゲノムデーターベースに存在しているIS(sll1474)が実験に用いたパスツールカルチャーコレクションの元株には存在していなかった。その結果、配列決定された株ではISの前後にある独立したORFとして命名されていたsll1473,sll1475はパスツールカルチャーコレクションの元株では一つのORF(sll1473-sll1475)であり光受容タンパク質のフィトクローム様の分子として機能している可能性が示唆された。さらにsll1474をプローブとしてサザンブロット解析を行ったところ合計4つのISが配列決定株でのみ挿入されていることを明らかにした。 2)運動関与遺伝子の検索と破壊とその表現形 バクテリアの運動に関係すると思われる遺伝子のSynechocystis sp.PCC6803のデーターベース上から相同遺伝子を検索した。検索結果から8種類の候補遺伝子を選択し、それらの遺伝子の破壊株を作製した。これら8種類の候補遺伝子のなかでpilT1(slr0161)の破壊株において寒天プレート上でコロニーが拡がらなくなるという表現形が観察された。この表現形は運動能が失われた結果であると考え、pilT1破壊株の解析を行った。pil遺伝子群は、Pseudomonas,Myxococcus,Neisseria,E.coli,など広い範囲のバクテリアに存在するピリ(pili)と呼ばれる線毛の生合成関与する遺伝子群である。 ネガティブ染色法による電子顕微鏡観察により、野生株のSynechocystis sp.PCC6803の細胞表層の線毛は直径約4nm、長さ約2〜6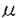 mの線毛が細胞から放射状にのびているのが観察された。一方pilT変異株にも線毛は存在した。また、その直径や形状に変化は認められなかった。多くの細胞を観察したところ、線毛の長さが伸長していることが明らかになった。 mの線毛が細胞から放射状にのびているのが観察された。一方pilT変異株にも線毛は存在した。また、その直径や形状に変化は認められなかった。多くの細胞を観察したところ、線毛の長さが伸長していることが明らかになった。 pilT1遺伝子はBacillusのDNA受容能に関わる、comG1遺伝子と約20%の相同性をもつ。BacillusではcomG1遺伝子が破壊されると外来のDNAを細胞内に取り込めなくなる。そこでPCC6803のpilT1破壊株においても形質転換効率を調べた。その結果、実験を行った条件下ではpilT1破壊株において形質転換体は得られず、野生株のおよそ100分の1以下にまで形質転換効率が低下していることが明らかになった。このことから、pilT1はPCC6803の形質転換にも必須な遺伝子であることが明らかになった。 3)pilT1遺伝子産物の機能 PilT1は中央部分にATP/GTP結合モチーフであるWalker box Aとバクテリアでのタンパク質輸送に関与するタンパク質、GPS(General Secretion Pathway)タンパク質のコンセンサス領域をもっている。以上のことから、PilT1はATPを基質として加水分解する際に得られるエネルギーで、一種の分子モーターとして働き細胞を運動させるのではないかと考えた。そこで大腸菌内でPilT1をHis-Tag標識した組み替え型タンパク質として発現させて、精製しATPase活性を測定した。その結果、精製したPilTの画分にATPase活性があることが明らかになった。抗pilT1抗体を用いたウエスタンブロット解析からPilT1は膜画分に存在することも明らかになった。 以上のように、申請者はいままでメカニズムの不明だったラン藻の運動に関して分子生物学的な手法により、解析の端緒をひらいた。この研究はラン藻のみにとどまらず、生物の運動能獲得という根元的なテーマに迫ることが期待される。さらに、この研究の過程で生理学、分子生物学の手法、及び科学的な方法論を広く修得した。よって、申請者は博士(学術)の学位を授与されるに値する。 |