大腸菌ファージP1由来の部位特異的リコンビナーゼCreは、34bPの認識配列(loxP)を特異的に認識し、相同組換えを行うための一連の反応をCre単独で行うことが知られている。この場合、同じ方向にloxP配列が配置されているとCreによりloxP配列に挟まれた領域が環状に切り出され、残りのゲノムはloxPを1分子残した状態で再結合する。近年、部位特異的リコンビナーゼを応用して、目的遺伝子とプロモーターの間にloxP配列を両側に持つstuffer領域を配置して発現制御を行う試みがトランスジェニックマウスで報告され注目されている。培養細胞においても部位特異的リコンビナーゼを用いた発現制御系の試みが行われているが、従来リコンビナーゼ遺伝子はトランスフェクション法で導入されていたため、100%の細胞での部位特異的相同組換えは不可能であった。 本研究では、Cre導入法としてアデノウイルスベクターを応用し、100%の細胞での発現ON制御を検討した。アデノウイルスベクターは、各種細胞へ効率よく遺伝子を導入することが可能であり、特に神経や筋肉などの静止期にある細胞への遺伝子導入も報告されている。また本研究で用いているアデノウイルスベクターは、ウイルスの増殖に必須であるE1A・E1B領域を欠損しているため、E1A・E1Bを恒常的に発現している293細胞以外ではウイルスが複製しない非増殖型であり、ベクターそのものによる細胞への影響は最小限であると考えられている。これらの特徴から、近年アデノウイルスベクターは基礎研究のみならず遺伝子治療用のベクターとしても注目されている。本研究に先立ち、発現のON制御後各種細胞で強力なプロモーターを検索したところ、CAGプロモーター(Cyto megalovirus enhancer. chicken 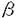 -actin promoter,rabbit -actin promoter,rabbit 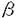 -globin polyA signalの複合型プロモーター)が、細胞種を問わず強力であることが示されたたため、CAGプロモーターの下流に発現制御用の配列を挿入すれば、発現ON制御により強力なプロモーターから100%の細胞で目的遺伝子を発現することが可能となることが想定された。 -globin polyA signalの複合型プロモーター)が、細胞種を問わず強力であることが示されたたため、CAGプロモーターの下流に発現制御用の配列を挿入すれば、発現ON制御により強力なプロモーターから100%の細胞で目的遺伝子を発現することが可能となることが想定された。 そこで、CAGプロモーターの支配下で、CreのN末端側にSV40ウイルスの核局在配列(NLS)を有する組換えアデノウイルス(AxCANCre:Cre発現ウイルス)を作製した。また、Creの標的ウイルスとして、CAGプロモーターの支配下にstufferとしてneo耐性遺伝子を有し、その下流にLacZ遺伝子を有する組換えアデノウイルス(Ax1CALNLZ)を作製した。Cre発現ウイルスと標的ウイルスを、ヒト及びサル由来の培養細胞へ同時感染し、stufferの遺伝子が環状に切り出された結果CAGプロモーターによって発現する目的遺伝子LacZ遺伝子の発現を、x-gal染色、ONPGによる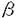 -galactosidase活性測定あるいはSouthern法による遺伝子解析により検討した(図1)。Cre発現ウイルスのコントロールウイルスには、CAGプロモーターとPolyAシグナルのみを有するAdex1CAwtを用いた。 -galactosidase活性測定あるいはSouthern法による遺伝子解析により検討した(図1)。Cre発現ウイルスのコントロールウイルスには、CAGプロモーターとPolyAシグナルのみを有するAdex1CAwtを用いた。 まず、CV1細胞へCre発現ウイルスをMOI 0.1から5、標的ウィルスをMOI 2.5で同時感染したところ、Cre発現ウイルスの量に依存して目的遺伝子であるLacZが発現し(図2)、MOI 2.5では100%の細胞がx-galで染色された。一方コントロールウイルスと標的ウイルスを同じMOIで感染した場合にもわずかの細胞が染色された。この結果については現在検討中であるが、今回観察されたバックグラウンドは、染色される細胞数ではCre発現ウイルスを感染した場合の1万分の1以下、ONPGによる活性測定では検出限界以下であった。 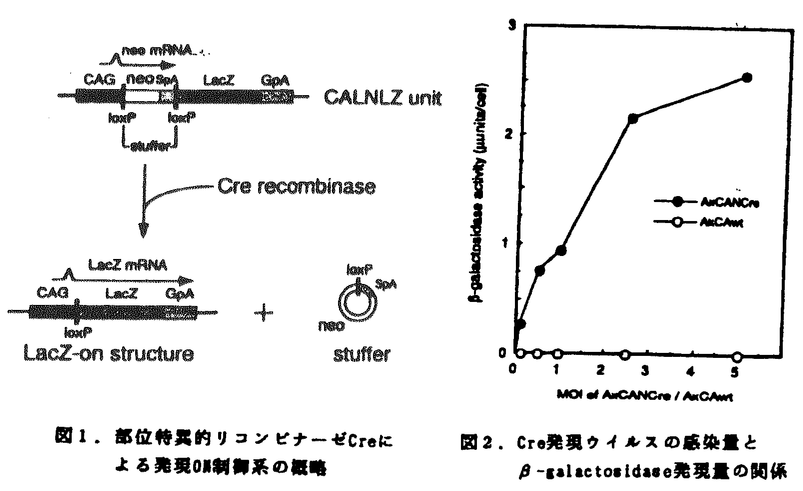 図表図1.部位特異的リコンビナーゼCreによる発現ON制御系の概略 / 図2.Cre発現ウイルスの感染量と 図表図1.部位特異的リコンビナーゼCreによる発現ON制御系の概略 / 図2.Cre発現ウイルスの感染量と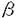 -galactosidase発現量の関係 -galactosidase発現量の関係 ここで認めらたLacZ遺伝子の発現が、CreによりloxP配列に挟まれたstuffer領域が環状に切り出された結果、CAGプロモーターからの目的遺伝子の発現がONになったことを遺伝子レベルで解析するために、CV1細胞へCre発現ウイルスをMOI 10、標的ウイルスをMOI 5で同時感染し、3日後の細胞からDNAを調製し、Southern法を用いて解析した。プローブにはstuffer-LacZの領域を用いた。その結果、Cre発現ウイルスを感染した場合のみ、stuffer領域が環状に切り出されていたことが明かとなり、先の染色の結果とも一致した。 この系が他の細胞においても機能することを確認するために、接着系の細胞としてHeLa、CV1細胞、血球系の細胞を代表して比較的アデノウイルスの感染効率の高いJurkat細胞を用いて検討した。HeLa及びCV1細胞へは、Cre発現ウイルスMOI 10、標的ウイルスをMOI 0.1から10で、Jurkat細胞へは、Cre発現ウイルスMOI 100、標的ウイルスMOI 1から100で同時感染した。その結果、HeLa、CV1細胞ではMOI 5まで、またJurkat細胞ではMOI 100までCre発現ウイルス量に依存して、LacZ遺伝子の発現量が増加しており、x-gal染色により100%の細胞が青く染色されていた(図3)。 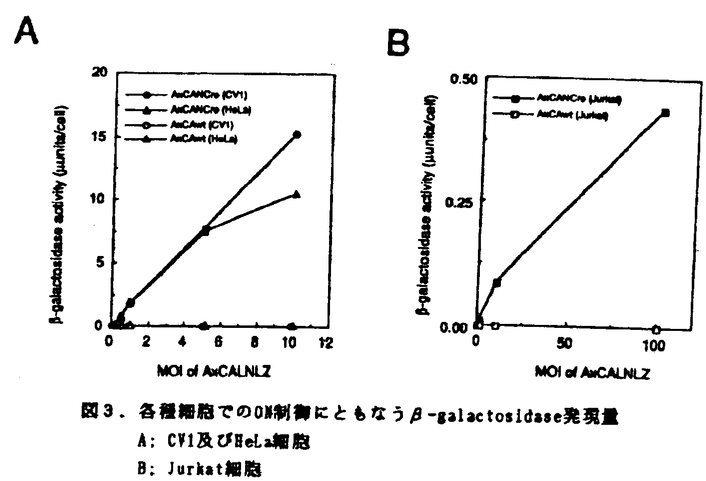 図3.各種細胞でのON制御にともなう 図3.各種細胞でのON制御にともなう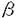 -galactosidase発現量A:CV1及びHeLa細胞 B:Jurkat細胞 -galactosidase発現量A:CV1及びHeLa細胞 B:Jurkat細胞 以上の結果から、Cre発現ウイルスによりstuffer領域を環状に切り出すことで、100%の細胞においてstufferであるneo耐性遺伝子を環状に切り出し、その下流のLacZ遺伝子の発現をONに調節できたことが明かとなった。 今回の研究から、部位特異的リコンビナーゼCreの系と組換えアデノウイルスの系を併用することにより、100%の細胞における比較的精度の高い発現調節系が確立された。従来の誘導型プロモーターは、誘導前のバックグラウンドが高く、しかも誘導後の発現は用いたプロモーターの強度に依存しているため、精度、強度の両立は困難であった。今回確立したCre発現組換えアデノウイルスによる発現調節系は、誘導前のバックグラウンドは低く、しかも誘導後は大変強力なCAGプロモーターによって目的遺伝子を発現することが可能である。 本論文では示していないが、組換えアデノウイルスを用いたCreによる発現制御系は、細胞の染色体上にある標的配列に対しても有効である。本法を用いることにより、細胞を樹立化する前に標的配列を有する組換えアデノウイルスを用いて、Creによる発現制御系が目的とする細胞で可能か否かを検討することが可能となった。また、組換えアデノウイルスの作製が困難である遺伝子を100%の細胞に導入したい場合には、発現OFFの型で組換えアデノウイルスを作製後、Cre発現ウイルスと同時感染することにより、目的遺伝子の発現をONにする系としても有用であると考える。 既に共同研究者により、この組換えアデノウイルスはES細胞やマウスの受精卵へも遺伝子導入が可能であることが報告されている。将来的にはトランスジェニックマウス作製の際の遺伝子発現制御や、遺伝子治療の際の過剰発現のOFF制御等への応用も期待できると考えている。 |